Entwicklung von Dithiocarbamaten als Anthelminthika und Hemmstoffen der RNA-Helikase eIF4A als potentielle antivirale Wirkstoffe
Projektbeschreibung
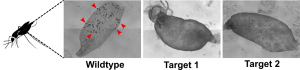
Wurmerkrankungen stellen ein großes Problem in großen Teilen der Welt dar. Verglichen mit der Krankheitslast ist die Zahl der zur Verfügung stehenden Wirkstoffe gering. In zahlreichen aufeinanderfolgenden Zyklen von Design, Synthese und Testung soll die neue Wirkstoffklasse der Dithiocarbamate hinsichtlich ihrer Wirkung an verschiedenen Helminthen, ihrer Wirtstoxizität und ihrer generellen Wirkstoffeigenschaften optimiert werden. Derivate werden synthetisiert, auf ihre Wirkung auf verschiedene pathogene Würmer und ihre Toxizität gegen humane Zellen getestet. Von vielversprechenden Derivaten werden ADME-Parameter bestimmt und einzelne Verbindungen ggf. einer in-vivo Testung zugeführt.
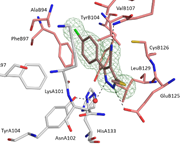
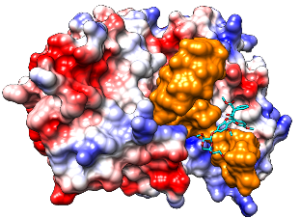
Diverse humanpathogen Viren, u.a. auch das Ebola- und das SARS-CoV-2-Virus, sind für ihre intrazelluläre Vermehrung auf Wirtsfaktoren angewiesen, die damit geeignete Zielstrukturen für die Entwicklung anti-viraler Wirkstoffe darstellen. Einer dieser Wirtsfaktoren ist die RNA-Helicase eIF4A. Basierend auf der Kristallstruktur werden potentielle Liganden konstruiert, in den Protein-RNA-Komplex gedockt und bei guter Passung synthetisch hergestellt. Die Verbindungen werden auf ihre Wirkung hinsichtlich der Translationseffizienz getestet. Gut wirksame Derivate werden anschließend auf ihren Hemmeffekt auf die Virusvermehrung in Zellkulturen getestet.

Wissenschaftliches Ziel:
Ziel in beiden Projekten ist es, Wirkstoffmoleküle zu entwickeln, die die Qualität einer Leitstruktur oder sogar eines Entwicklungskandidaten aufweisen.
DRUID-Kooperationspartner:
A2, A3, A4, A7, B2, B7P, C6 NWG, D4, E1, E4, E6
Literatur B5: –