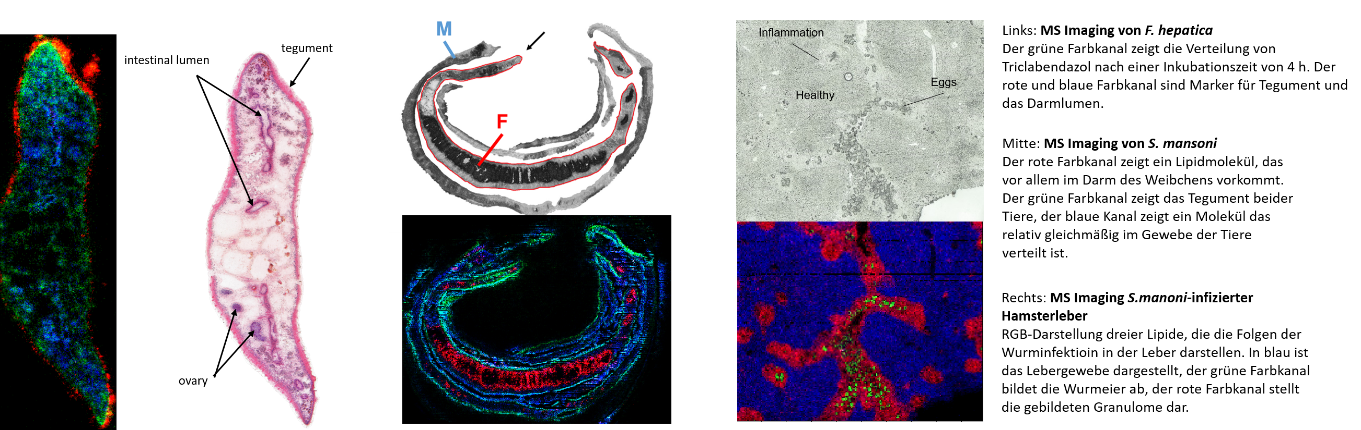
Prozessentwicklung/-kontrolle (PAT, cGMP), Plattformen für die Produktion von Proteinen und Virusähnlichen Partikeln
Projektbeschreibung
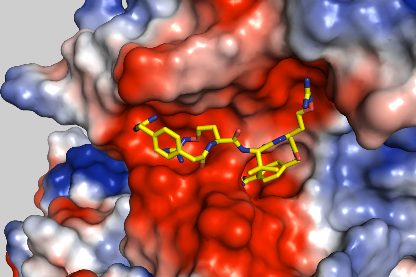
Um Forschungsergebnisse aus dem DRUID Konsortium in die Klinik oder die Industrie zu translatieren, sind robuste Produktionsprozesse, die mehr als eine bloße Maßstabsvergrößerung darstellen, unabdingbar. Dabei müssen diese Prozesse den Richtlinien der guten Herstellungspraxis (cGMP) und der Prozessanalytischen Technologie (PAT) folgen. Folgende Arbeiten sind geplant: i) Prozessintensivierung und Ausbau der BEVS-Produktionsplattform sowie Umsetzung einer kontinuierlichen Prozessführung, ii) Einbindung von online PAT-Technologie, z.B. Impedanzspektroskopie zur automatischen Bestimmung des optimalen Erntezeitpunkts und der automatisierten Ernte, iii) Expression (BEVS) und Aufreinigung von S. mansoni-Kinasen sowie Untersuchung der Kinasen und putativer Inhibitoren, und iv) Untersuchung neuartiger Transfektionsreagenzien für die transiente Proteinproduktion im Bioreaktormaßstab.
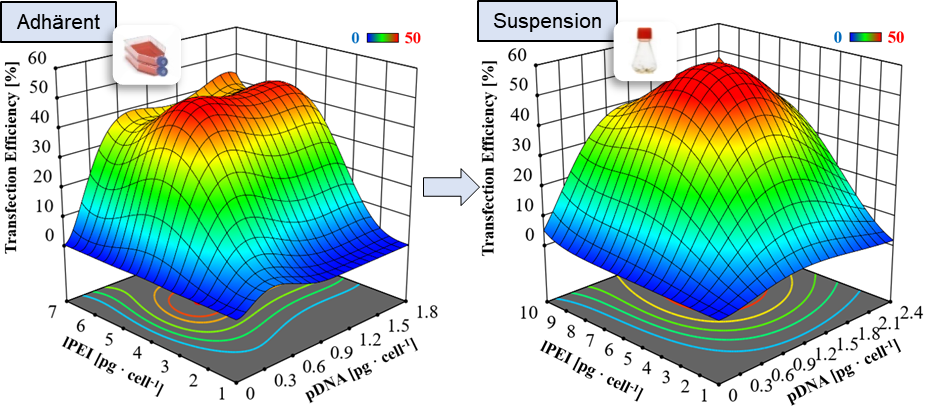
Optimierung des transienten Transfektionsprozesses mittels statistischer Versuchsplanung. ©IBPT
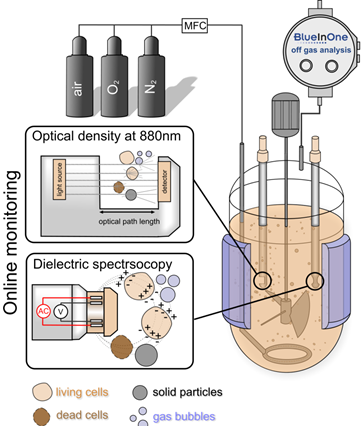
Produktionskonzept mit integrierter PAT-Technologie. ©IBPT
Wissenschaftliches Ziel:
Die Arbeiten zur Produktionsprozessentwicklung und Prozessanalytik werden weiter an die Fragestellungen des Zentrums angepasst, erweitert, und dem gesamten Konsortium zur Nutzung angeboten. Insbesondere ist geplant, die aufgebaute BEVS (Baculovirus-Expression-System)-Plattform weiter zu intensivieren und zu automatisieren. Darüber hinaus wird auch die zweite Produktionsplattform – die transiente Produktion mit HEK-293T-Zellen – weiterentwickelt.
DRUID-Kooperationspartner:
B4 AG Schlitzer, B5 AG Grevelding, B7 P AG Falcone, C6 NWG Häberlein, E1 Plattform Grevelding/Häberlein
Literatur E5: Biotechnol, DOI: 10.1016/j.ejbt.2021.08.002, 3. Lothert et al. (2020) Methods Mol Biol 2183:217-248, 4. Dekevic et al. (2022) J Biotechn 346 23-34, 5. Schwarz et al. (2021) Elec J Biotechnol, DOI: 10.1016/j.ejbt.2022.01.003; 6. Barekzai et al. (2020) New Adv Ferm Processes, DOI: 10.5772/intechopen.90029, 7. Eckhardt et al. (2021) Sep Sci Technol, 57 (6) 886-897