Projektbereich B:
B1Dengue- und Zikavirus, Design von Inhibitoren der NS3/NS2B-Serinprotease
Wibke Diederich, Peter Kolb
Prof. Dr. Wibke Diederich
Institut für Pharmazeutische Chemie
Zentrum für Tumor- und Immunbiologie
Philipps-Universität Marburg
Hans-Meerwein-Straße 3
35043 Marburg
Tel.: +49 (0)6421-28 25810
Fax: +49 (0)6421-28 26254
E-Mail: wibke.diederich(at)staff.uni-marburg(dot)de
Prof. Dr. Peter Kolb
Institut für Pharmazeutische Chemie
Philipps-Universität Marburg
Marbacher Weg 8
35032 Marburg
Tel.: +49 (0)6421-28 25908
Fax: +49 (0)6421-28 26652
E-Mail: peter.kolb(at)uni-marburg(dot)de
Projektbeschreibung
Infektionen mit dem Dengue-Virus haben in den letzten Jahren mit rund 50-100 Millionen Neuinfektionen pro Jahr einen neuen Höchststand erreicht. In der überwiegenden Zahl der Fälle verläuft die Infektion mit milden, grippeähnlichen Symptomen, jedoch entwickelt ein kleiner Prozentsatz der Betroffenen, häufig Kinder, ein hämorrhagisches Fieber, das bei schwerem Verlauf tödlich verläuft. Zwar steht inzwischen ein Impfstoff zur Verfügung, da dieser aber nur für einen sehr begrenzten Personenkreis zugelassen ist, ist die Entwicklung von Wirkstoffen, die die Vermehrung des Virus effizient unterdrücken, unabdingbar. Im Fokus unser Arbeiten steht die viruseigene Serinprotease NS3/NS2B, die das virale Vorläuferprotein in funktionelle Proteine spaltet und für Reifung des Virus unerlässlich ist.
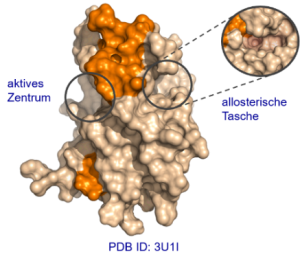
Abb. B1. Kristallstruktur der NS3/NS2B Protease DENV 3.1
Wissenschaftliches Ziel:
Ziel des Projektes ist die Weiterentwicklung allosterischer Inhibitoren der viruseigenen Serinprotease NS3/NS2B, nicht nur hinsichtlich ihrer Affinität, sondern auch in Bezug auf ihre pharmakokinetischen Eigenschaften und ihre Toxizität (hit-to-lead-Entwicklung) mittels eines kombinierten Ansatzes aus computergestütztem Design, Synthese, biologischen Tests und der Kristallstrukturanalyse. Darüber hinaus sollen basierend auf den gewonnenen Erkenntnisse Inhibitoren der verwandten Zika-Protease entwickelt werden.
DRUID-Kooperationspartner:
A1 Becker, A2 Grünweller, B3 Rahlfs/Kolb/van Zandbergen, XY Herker, E6 Schiffmann/Laux
Literatur B1: [1] Noble et al. (2012) J Virol 86(1):438-446; *[2] Chevillard et al. (2015), J Chem Inf Model 55(9):1824-1835; *[3] Chevillard et al. (2018) J Med Chem, 61(3):1118-1129.
B2Virale Protein-de-ADP-Ribosylierungs- und NMPylierungs-Aktivitäten als potentielle therapeutische Angriffspunkte gegen Coronaviren
John Ziebuhr
Prof. Dr. John Ziebuhr
Institut für Medizinische Virologie
Biomedizinisches Forschungszentrum
Seltersberg (BFS)
Justus-Liebig-Universität Gießen
Schubertstraße 81
35392 Gießen
Tel.: +49 (0)641-99 41200
Fax: +49 (0)641-99 41209
E-Mail: John.Ziebuhr(at)viro.med.uni-giessen(dot)de
Projektbeschreibung
Coronaviren sind wichtige Pathogene von Mensch und Tier und verursachen v. a. respiratorische und intestinale Infektionen. Sie besitzen ein hohes zoonotisches Potential und haben wiederholt zu Ausbrüchen schwerer respiratorischer Infektionen beim Menschen geführt, wie die seit 2019 laufende SARS-CoV-2-Pandemie eindrücklich belegt. Die zur Verfügung stehenden therapeutischen Möglichkeiten für schwere Verlaufsformen von COVID-19 und anderen Coronavirusinfektionen sind extrem begrenzt, so dass die Entwicklung neuer Therapeutika eine hohe Priorität besitzt. Das Forschungsprojekt B2 konzentriert sich auf zwei coronavirale Proteine, die bei allen bekannten Coronaviren konserviert sind: die Makrodomäne (macD) im Nichtstrukturprotein 3 (nsp3) sowie eine neu entdeckte Nukleotidyltransferase (NiRAN), die mit der viralen RNA-Polymerase im nsp12 verbunden ist und für die Virusvermehrung essentiell ist.
Coronavirales Replikase-Polyprotein 1ab, aus dem durch proteolytische Spaltung (PL1, PL2, 3CL) 16 Nichtstrukturproteine mit zahlreichen enzymatischen und anderen Funktionen freigesetzt werden.
Wissenschaftliches Ziel:
Das Projekt zielt darauf ab, die biochemischen Eigenschaften der beiden Proteine umfassend zu charakterisieren und ihre Funktionen im viralen Replikationszyklus in geeigneten Zellkulturmodellen zu bestimmen. Auf Grundlage der konservierten Substratspezifität der NiRAN-Domäne sollen HTS-Assays entwickelt und zur Identifizierung potentieller Inhibitoren genutzt werden.
DRUID-Kooperationspartner:
A2 Grünweller, A3 Weber, C5 Kraiczy, E3 Rahlfs/Przyborski
Literatur B2: 1. *Slanina et al. (2021) Proc Natl Acad Sci USA 118: e2022310118 2. *Krichel et al. (2021) Sci. Adv. 7: eabf1004 3. *Müller et al. (2021) Antiviral Res 175: 1004706 4. *Snijder et al. (2016) Adv Virus Res 96: 59-126 5. *Putics et al. (2005) J Virol 79: 12721-31.
B3NAD(P)H-abhängige Stoffwechselwege als Ziele für neue Antiinfektiva
Stefan Rahlfs, Ger van Zandbergen, Peter Kolb
Dr. Stefan Rahlfs
Biochemie und Molekularbiologie
Justus-Liebig-Universität Gießen
Heinrich-Buff-Ring 26-32
35390 Gießen
Tel.: +49 (0)641-99 39117
E-Mail: stefan.rahlfs(at)ernaehrung.uni-giessen(dot)de
Prof. Dr. Ger van Zandbergen
Abteilung Immunologie
Paul-Ehrlich-Institut
Paul-Ehrlich-Str. 51-59
63225 Langen
Tel.: +49 (0)6103-77 2005
E-Mail: Ger.Zandbergen(at)pei(dot)de
Prof. Dr. Peter Kolb
Institut für Pharmazeutische Chemie
Philipps-Universität Marburg
Marbacher Weg 8
35032 Marburg
Tel.: +49 (0)6421-28 25908
Fax: +49 (0)6421-28 26652
E-Mail: peter.kolb(at)uni-marburg(dot)de
Projektbeschreibung
Das zelluläre Redoxgleichgewicht spielt für pathogene Mikroorganismen eine essentielle Rolle. Zentral daran beteiligt sind Enzyme des NAD(P)H-abhängigen Glutathion- und Thioredoxinsystems1,2, sowie die Glucose-6-Phosphatdehydrogenase (G6PD) und 6-Phosphogluconatdehydrogenase (6PGD), die maßgeblich zur Bereitstellung von NADPH und Ribose-5-Phosphat über den Pentosephosphatweg beitragen3. Die bifunktionale G6PD der Malariaparasiten Plasmodium falciparum und P. vivax (GluPho) unterscheidet sich funktionell und strukturell von den humanen Wirtsenzymen4 und ist essenziell für den Malariaparasiten5. In Kooperation mit dem Sanford-Burnham Institute/UCSD, La Jolla, konnten wir für die PfGluPho einen high-throughput kompatiblen Assay etablieren und ca. 400.000 Substanzen (u.a. NIH MLSMR Collection) screenen6. Anschließende Struktur-Wirkungs-Analysen und Lead-Optimierung resultierten in den im nanomolaren Bereich aktiven und hoch selektiven PfGluPho Inhibitor SBI-7507. Das Konzept konnte bereits auf Leishmania übertragen werden, die 3D-Kristallstruktur der Leishmania donovani G6PD und PGD gelöst werden und auf deren Basis ein erstes in silico Screening von Kleinmolekülen durchgeführt werden. Darüber hinaus wurde ein high-throughput kompatibler Assay für die rekombinanten G6PDs und 6PGDs aus Leishmania etabliert, um ein Inhibitor-Screening bei dem Novartis FAST lab in Cambridge (USA) durchzuführen.
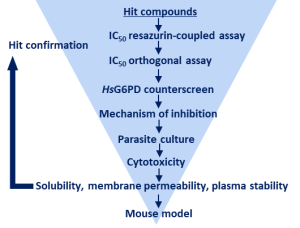
Workflow-Diagramm zur Inhibitor Identifizierung gegen G6PDs/PGDs
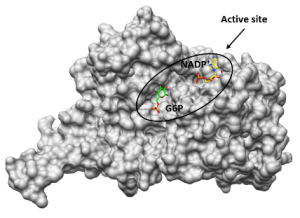
3D-Kristallstruktur LdG6PD ©Isabell Berneburg
Wissenschaftliches Ziel:
Ziel des Projektes ist die funktionelle und strukturelle Charakterisierung der Enzyme G6PD und 6PGD aus Leishmania und Plasmodium als Zielmoleküle für die Wirkstoffentwicklung, sowie die Identifizierung und Weiterentwicklung von Inhibitoren dieser Enzyme (in silico und HTS). Das Konzept soll auch auf weitere Erreger innerhalb des DRUID Konsortiums, wie z.B. Schistosoma, übertragen werden.
DRUID-Kooperationspartner:
B1 Diederich/Kolb; B4 Grevelding; D3 van Zandbergen; E1 Grevelding/Häberlein; E3 Rahlfs/Przyborski; E4 Spengler; E6 Schiffmann/Laux
Literatur B3: 1. Fritz-Wolf et al. (2011) Nature Comm. 2:383* 2. Koncarevic et al. (2009) PNAS 106: 13323-8* 3. Bozdech and Ginsburg (2005) Malaria J 3:23 4. Jortzik et al. (2011) Biochem J Energy 436:641-50* 5. Allen et al. (2015) FEBS J 282:3808-23*, 6. Preuss et al. (2012) J Med Chem 55:7262-72* 7. Berneburg et al. (2022) Antimicrob Agents Chemother (accepted)*
*eigene Publikationen
B4Schistosoma mansoni: Biologische Zielstrukturen und Inhibitorentwicklung
Christoph Grevelding
Prof. Dr. Christoph Grevelding
BFS, Institut für Parasitologie
Justus-Liebig-Universität Gießen
Schubertstraße 81
35392 Gießen
Tel.: +49 (0)641-99 38466
Fax: +49 (0)641-99 38469
E-Mail: Christoph.Grevelding(at)vetmed.uni-giessen(dot)de
Projektbeschreibung
Hintergrund: Beim dem human- und tierpathogenen Wurmparasiten Schistosoma mansoni hängt die sexuelle Reifung des Weibchens von einem kontinuierlichen Paarungskontakt mit dem Männchen ab (Abb. 1). Die Paarung ist somit auch für die Produktion von Wurmeiern und der daraus resultierenden Pathogenese der Schistosomiasis entschei-dend. Wir haben gezeigt, dass verschiedene Molekülklassen in diese Reproduktionsprozesse involviert sind.1 So offenbarten Versuche mit Kinaseinhibitoren und RNA-Interferenz gegen Kinasen, dass diese Moleküle Zellteilungsaktivität, Spermatogenese, Oogenese, Eiproduk-tion und Vitalität von Würmern in vitro negativ beeinflussen. Der Abl-TK-Inhibitor Imatinib (Krebsmedikament Glivec) induzierte in adulten S. mansoni eine gestörte Reproduktion sowie die Ablösung der Gastro-dermis mit letalen Folgen.2,3
Neben Kinasen, die wir in Kooperation mit der AG Falcone bearbeiten4, stehen weitere Enzyme im Fokus unserer Untersuchungen. Dazu zählen Aldehyddehydrogenasen (ALDH) und Aldehydreduktasen (AR), die durch molekularen Stress induzierte Prozesse in Schistosomen regulieren. Ihre Inhibition könnte den Parasiten schwächen. Erste in vitro-Experimente mit Inhibitoren gegen diese Moleküle, wie z.B. dem ALDH-Inhibitor Disulfiram, führten in kurzer Zeit zu morphologischen Veränderungen, Entpaarung, reduzierter Vitalität sowie zum Tod des Parasiten in vitro. Aber auch Helikasen können interessante Angriffspunkte darstellen, wie Arbeiten mit dem RNA-Helikase-Inhibitor Silvestrol gezeigt haben.5 In adulten S. mansoni reduzierte Silvestrol die Fitness adulter Würmer in vitro und die Stammzellproliferation in Keimzellen (Abb. 2). Mit den AGs Prof. Schlitzer und Prof. Grünweller (Marburg) werden wir an diesen Zielmolekülen arbeiten und an der Entwicklung synthetischer Inhibitoren6,7, um physiologische und morphologische Effekte in vitro und bei Topkandidaten dann im Infektionsmodell in vivo zu untersuchen.

Abb. 1. Lichtmikroskopische Aufnahme eines Schistosomen-Pärchens in vitro. Während des dauerhaften Paarungskontaktes liegt das Weibchen (Pfeil) in einer Bauchfalte des Männ-chens. Das ist eine wesentliche Voraussetzung für die Eiproduk-tion (Sterne).
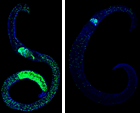
Abb. 2. EdU-Färbung von S. mansoni-Weibchen vor (links) und nach Silvestrol-Behandlung (rechts). Die Anzahl der Stammzellen (grün) ist signifikant redu-ziert (rechts).
Wissenschaftliches Ziel:
Wir werden die Klonierung sowie die molekulare und biochemische Charakterisierungen zweier RNA-Helikasen aus S. mansoni durchführen. Unterstützt durch verschiedene Kooperationspartner/innen im DRUID-Konsortium (s.u.) werden wir die rekombinanten Proteine in Enzymassays gegen Silvestrol und synthetische Derivate testen. Ferner planen wir, mit den bereits rekombinant hergestellten Enzymen SmALDH312 und SmAR, weitere potenzielle Zielmoleküle, Enzymtests mit Inhibitoren durchzuführen. Hierzu zählen u.a. Disulfiram-derivate, die bereits anti-schistosomale Effekte in vitro gezeigt hatten. Experimenten zur Kristallisationen und Strukturaufklärungen dieser Moleküle werden auch durchgeführt. Ziel ist die Entwicklung von Wirkstoffen mit inhibitorischer Aktivität gegen die ausgewählten Zielproteine, hoher Spezifität und Bioverfügbarkeit bei gleichzeitig reduzierter Toxizität.8 In vitro– und später auch in vivo-Experimente sollen die Übertragbarkeit der gewonnenen Erkenntnisse auch auf andere Parasiten zeigen (Plattformprojekt E1).
DRUID-Kooperationspartner:
A2 AG Grünweller, B3 AG Rahlfs/Kolb/van Zandbergen, B5 AG Schlitzer, E1 Platformprojekt/Häberlein, E3 AG Rahlfs/Przyborski, E5 AG Czermak/Salzig lab
Literatur B4: 1Beckmann et al. (2010) PLoS Pathog 6:e1000769; 2Beckmann et al. (2010) Int J Parasitol 40:521-6; 3Beckmann et al. (2012) Curr Pharm Des 18:3579-94; 4Moreira et al. (2022) Molecules (in press); 5Taroncher-Oldenburg et al. (2021) Microorganisms 9(3):540; 6Peter Ventura et al. (2021) ChemMedChem 14(21):1856-62; 7Peter Ventura et al. (2012) Arch Pharm (Weinheim) 354(12):
B5Entwicklung von Dithiocarbamaten als Anthelminthika und Hemmstoffen der RNA-Helikase eIF4A als potentielle antivirale Wirkstoffe
Martin Schlitzer
Prof. Dr. Martin Schlitzer
Institut für Pharmazeutische Chemie
Fachbereich Pharmazie
Philipps-Universität Marburg
Marbacher Weg 6
35037 Marburg
Tel.: +49 (0)6421 28-25840
E-Mail: schlitzer(at)staff.uni-marburg(dot)de
Projektbeschreibung
Wurmerkrankungen stellen ein großes Problem in großen Teilen der Welt dar. Verglichen mit der Krankheitslast ist die Zahl der zur Verfügung stehenden Wirkstoffe gering. In zahlreichen aufeinanderfolgenden Zyklen von Design, Synthese und Testung soll die neue Wirkstoffklasse der Dithiocarbamate hinsichtlich ihrer Wirkung an verschiedenen Helminthen, ihrer Wirtstoxizität und ihrer generellen Wirkstoffeigenschaften optimiert werden. Derivate werden synthetisiert, auf ihre Wirkung auf verschiedene pathogene Würmer und ihre Toxizität gegen humane Zellen getestet. Von vielversprechenden Derivaten werden ADME-Parameter bestimmt und einzelne Verbindungen ggf. einer in-vivo Testung zugeführt.
Diverse humanpathogen Viren, u.a. auch das Ebola- und das SARS-CoV-2-Virus, sind für ihre intrazelluläre Vermehrung auf Wirtsfaktoren angewiesen, die damit geeignete Zielstrukturen für die Entwicklung anti-viraler Wirkstoffe darstellen. Einer dieser Wirtsfaktoren ist die RNA-Helicase eIF4A. Basierend auf der Kristallstruktur werden potentielle Liganden konstruiert, in den Protein-RNA-Komplex gedockt und bei guter Passung synthetisch hergestellt. Die Verbindungen werden auf ihre Wirkung hinsichtlich der Translationseffizienz getestet. Gut wirksame Derivate werden anschließend auf ihren Hemmeffekt auf die Virusvermehrung in Zellkulturen getestet.

Wissenschaftliches Ziel:
Ziel in beiden Projekten ist es, Wirkstoffmoleküle zu entwickeln, die die Qualität einer Leitstruktur oder sogar eines Entwicklungskandidaten aufweisen.
DRUID-Kooperationspartner:
A2, A3, A4, A7, B2, B7P, C6 NWG, D4, E1, E4, E6
Literatur B5: –
B6 PIdentifizierung von antiviralen Targets im Lipidmetabolismus
Eva Herker
Prof. Dr. Eva Herker
Institut für Virologie
Philipps-Universität Marburg
Hans-Meerwein-Str. 2
35043 Marburg
Tel.: +49 (0)6421-28-64525
E-Mail: eva.herker(at)uni-marburg(dot)de
Projektbeschreibung
Die humanpathogenen Flaviviren Dengue (DENV), Gelbfieber (YFV), Zika (ZIKV), West-Nil (WNV) und das Frühsommer-Meningoenzephalitisvirus (TBEV/FSME) verursachen akute Infektionen mit teils schweren Komplikationen. Fundamentale Schritte in der Flavivirusreplikation sind eng mit zellulären Lipiden verknüpft. Hierzu zählen unter anderem Membranreorganisationen zur Bildung der Replikationsvesikel oder die Bildung der Virushülle. Flaviviren verändern die Lipidkomposition der Wirtszelle zu ihrem Vorteil, und die Aktivität verschiedener lipidmetabolisierender Enzyme ist essentiell für die erfolgreiche Replikation. Aus diesem Grund stellen Enzyme verschiedener Lipidstoffwechselwege interessante Targets für eine (pan-) antiflavivirale Therapie dar. Allerdings ist noch nicht detailliert geklärt, ob die o.g. Vertreter der Flaviviren auf unterschiedliche oder ähnliche Zweige des Lipidmetabolismus angewiesen sind.
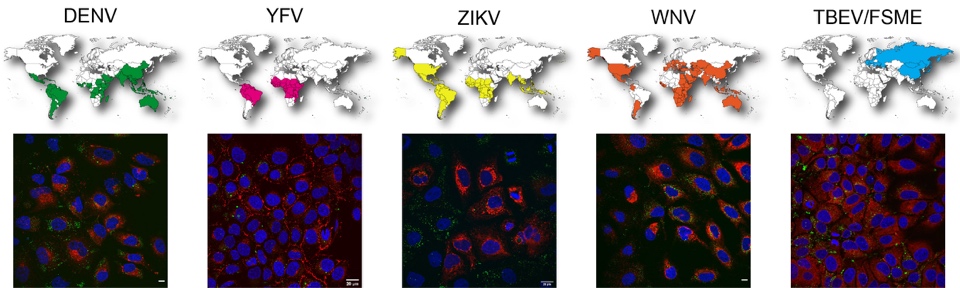
Immunfluoreszenzmikroskopie von Zellen, die mit verschiedenen Flaviviren infiziert sind; rot: virales E-Protein; grün: lipid droplet
Wissenschaftliches Ziel:
Das Projekt zielt darauf ab, antivirale Angriffspunkte im zellulären Lipidmetabolismus zu identifizieren. Hierbei wird die Rolle verschiedener Schlüsselenzyme der de novo Fettsäure- und Cholesterol-Biosynthese, des Phospholipid- und Neutrallipidstoffwechsels, sowie von Enzymen der Lipidremodellierung in der Flavivirusreplikation mittels eines shRNA-basierten Screenings analysiert. Zusätzlich werden verschiedene Inhibitoren getestet. Die Ergebnisse werden in verschiedenen Zelltypen validiert und die molekularen Mechanismen detailliert untersucht.
DRUID-Kooperationspartner:
B1 Diederich/Kolb, D1 Böttcher-Friebertshäuser/Steinmetzer, E7P Krijnse-Locker, E4 Spengler
Literatur B6P: 1. Herker et al. (2010) Nat Med 16: 1295 2. Harris et al. (2011) J Biol Chem 286: 42615 3. Herker et al. (2012) J Biol Chem 287: 2280 4. Rosch et al. (2016) Cell Rep 16: 3219 5. Hofmann et al. (2018) Biochim Biophys Acta Mol Cell Biol Lipids 1863: 1041 6. Schobel et al. (2018) Sci Rep 8: 3893 7. Lassen et al. (2019) J Cell Sci 132: jcs.217042 8. Bley et al. (2020) Int J Mol Sci 21: 9. Herker et al. (2021) Trends Cell Biol 31: 345 10. Nguyen-Dinh et al. (2021) Cells 10: 2407
B7PNeue Wege zur Kontrolle von Schistosomiasis und Echinokokkose
Franco Falcone
Prof. Dr. Franco Falcone
Institut für Parasitologie
BFS - Biomedizinisches Forschungszentrum Seltersberg
Justus-Liebig-Universität Gießen
Schubertstraße 81
35392 Gießen
Tel.: +49 (0)641-99 38030
E-Mail: franco.falcone(at)vetmed.uni-giessen(dot)de
Projektbeschreibung
Echinokokkose und Schistosomiasis sind zwei sogenannte vernachlässigte Tropenkrankheiten, die die Gesundheit der Betroffenen in endemischen Ländern stark beeinträchtigen. Unsere Gruppe entwickelt neue Instrumente, die für eine bessere Kontrolle dieser beiden Krankheiten eingesetzt werden können. Für Bilharziose entwickeln wir neue Medikamente, die auf die schistosomalen Mitogen-aktivierten Proteinkinasen abzielen [1], um neue Verbindungen zu finden, die sowohl larvizid als auch adultizid sind. Für die Echinokokkose entwickeln wir eine neuartige Diagnoseplattform, die auf dem Nachweis von parasitenspezifischem IgE unter Verwendung von humanisierten IgE-Reporterzelllinien beruht [2-4], die in verschiedenen Formaten eingesetzt werden können. Wir arbeiten auch an der Ausweitung dieser Diagnoseplattform auf andere parasitäre Infektionen (z. B. Fasciola, Zystizerkose, Clonorchis, Opisthorchis).
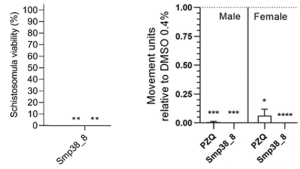
Larvizide und Adultizide Aktivität von Verbindung 38_8, die aus einer Substanzbibliothek mittels unseres bionformatischen Ansatzes ausgesucht wurde.
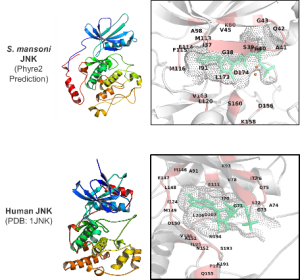
Strukturvorhersage von S. mansoni JNK im Vergleich zum humanen Orthologen
Wissenschaftliches Ziel:
Das Schistosomen-Projekt zielt darauf ab, mithilfe einer bioinformatischen in silico Analyse Leitmoleküle für Kinase-Inhibitoren zu identifizieren und diese zu geeigneten Arzneimitteln weiterzuentwickeln. Im Rahmen des Echinococcus-Projekts streben wir ein besseres Verständnis der schützenden Immunreaktionen gegen Echinokokken an und wollen dieses Wissen in die Entwicklung von Impfstoffen und stark verbesserten, innovativen Diagnosetechnologien einfließen lassen.
DRUID-Kooperationspartner:
A4 Heine lab, B4 Grevelding lab, C6 NWG Häberlein lab, E1 Czermak/Salzig lab
Literatur B7P: [1] *Pereira-Moreira et al. (2020) ACS Omega 5:9064-9070 [2] Kalli M et al. (2020) Sci Rep. 10:18208 [3] Kalli M. et al. (2020) Methods Mol Biol.;2163:155-162. [4] Prakash PS et al. (2021) Parasitol Res.








