A1Interaktionen von Ebola-Virus-Proteinen als Ziele antiviraler Strategien
Stephan Becker
Prof. Dr. Stephan Becker
Institut für Virologie
Philipps-Universität Marburg
Hans-Meerwein-Str. 2
35043 Marburg
Phone: +49 (0)6421-28 66253
E-Mail: becker(at)staff.uni-marburg(dot)de
Projektbeschreibung
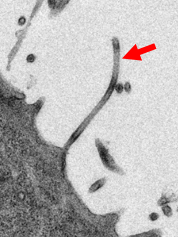
Elektronenmikroskopische Aufnahme von Ebolavirus infizierten Zellen. Die langgestreckten Viruspartikel werden von der Oberfläche der Zelle freigesetzt (roter Pfeil). ©Schauflinger
Das Ebolavirus (EBOV) verursacht schweres Fieber mit einer außerordentlich hohen Sterblichkeit. Das Matrixprotein VP40 von EBOV spielt eine Schlüsselrolle im viralen Replikationszyklus, der durch Homooligomerisierung des Proteins gesteuert wird. Die Dimerisierung von VP40 ist entscheidend für den Transport des Proteins zur Plasmamembran, wo die Dimere polymerisieren und zur Bildung von Filamenten führen, die die Knospung des Virus ermöglichen. Die VP40-Oktamerisierung führt zu einer Herunterregulierung der viralen RNA-Synthese. Aufgrund ihrer zentralen Rolle als Bausteine von Oligomeren höherer Ordnung stellen Dimere ein vielversprechendes Ziel für antivirale Medikamente dar. Mit Hilfe eines fragmentbasierten Ansatzes wurden kleine Moleküle identifiziert, die an VP40-Kristalle binden und zu Leitverbindungen weiterentwickelt werden sollen, um die VP40-Oligomerisierung und damit die Virusreplikation zu hemmen.
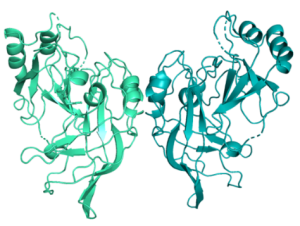
Hochauflösende Kristallstruktur des dimerischen Matrixproteins VP40 des Ebolavirus. ©Anke Werner
Wissenschaftliches Ziel:
Das Projekt zielt darauf ab, identifizierte Leitmoleküle zu antiviralen Medikamenten zu entwickeln, wobei strukturbasiertes Wirkstoffdesign – eine Kombination aus Proteinkristallographie und in silico-Methoden – sowie Zellkulturexperimente unter BSL4-Bedingungen zur Validierung eingesetzt werden.
DRUID-Kooperationspartner:
B1 Diederich/Kolb lab, A4 Heine/Reuter lab, D1 Steinmetzer lab, E3 Rahlfs/Przyborski lab
Literatur A1: 1. *Hartlieb et al. (2007) PNAS 104: 624-9 2. *Hartlieb et al., (2003) J. Biol. Chem. 278: 41830-6 3. *Hoenen et al. (2005) J Virol. 79: 1898-905 4. *Möller et al. 79, 14876-86 (2005) J Virol. 5. Hoenen et al. (2010) J Virol 84: 7053-63. 6. *Gomis-Ruth et al. (2003) Structure 11: 423-33.
A2Entwicklung von eIF4A-Inhibitoren als Kandidaten für eine klinische Anwendung sowie Charakterisierung von eIF4A-Varianten in Pathogenen
Arnold Grünweller
Prof. Dr. Arnold Grünweller
Philipps-Universität Marburg
Bau C
Marbacher Weg 6
35032 Marburg
Tel.: +49 (0)6421-2825849
Fax: +49 (0)6421-28 25854
E-Mail: arnold.gruenweller(at)staff.uni-marburg(dot)de
Projektbeschreibung
Die zelleigene RNA-Helikase eIF4A ist ein exzellentes Target für die Entwicklung antiviraler Breitbandmedikamente. Dieses Enzym wird für den Start der viralen Proteinsynthese von vielen Viren, insbesondere Coronaviren, benötigt und kann effizient und spezifisch durch die Stoffgruppe der Rocaglate gehemmt werden. In Projekt A2 möchten wir entsprechende Wirkstoffe für eine klinische Anwendung weiterentwickeln, indem wir unter anderem Rocaglate für eine lokale Applikation in den respiratorischen Trakt vernebeln und ein detailliertes Nebenwirkungsprofil erstellen. Zudem sollen neue eIF4A-Inhibitoren identifiziert und charakterisiert werden. Durch systematische Mutagenese bekannter Coronavirus-Sequenzen soll außerdem eine Vorhersage der Rocaglat-Sensitivität bei neu auftretenden Coronaviren ermöglicht werden. Schließlich soll die therapeutische Relevanz von Rocaglaten in verschiedenen DRUID-relevanten Pathogenen, die Varianten von eIF4A exprimieren, untersucht werden.
![]()

Festklemmen der RNA (RNA clamping) an der Oberfläche von eIF4A durch das Rocaglat Silvestrol sowie Nachweis der Hemmung der (corona)viralen Proteinsynthese durch das Rocaglat CR-31-B (-).
Wissenschaftliches Ziel:
Rocaglate sollen für eine klinische Anwendung weiterentwickelt und eine Vorhersage der Rocaglat-Sensitivität bei neu auftretenden Coronaviren und anderen DRUID-relevanten Pathogenen ermöglicht werden.
DRUID-Kooperationspartner:
B2 Ziebuhr lab, B4 Grevelding lab, B5 Schlitzer lab, C6 Häberlein lab, D1 Friebertshäuser/Steinmetzer lab, D3 van Zandbergen lab, D4 Hermosilla/Mazurek/Taubert lab, E4 Spengler lab, E6 Schiffmann lab
Literatur A2: 1.*Biedenkopf et al., (2017), Antiviral Res. 137: 76-81; 2. *Müller et al., (2018), Antiviral Res. 150:123-129; 3. *Elgner et al., (2018), Viruses. 10(4): 149; 4. *Glitscher et al., (2018), Viruses. 10(6): 301; 5. *Henß et al., (2018), Viruses. 10(11): 592; 6. *Müller et al., (2020), Antiviral Res. 175:104706; *7. *Müller et al., (2021), Antiviral Res. 186: 105012; 8. *Blum et al., (2020), J Cell Mol Med. 24(12): 6988-6999; [*eigene Publikationen].
A3Posttranslationale Proteinmodifikationen als Achillesferse pathogener RNA-Viren
Friedemann Weber
Prof. Dr. Friedemann Weber
Institut für Virologie
FB Veterinärmedizin
Justus-Liebig-Universität Gießen
Schubertstraße 81
35392 Gießen
Phone: +49 (0)641-99 38350
E-Mail: friedemann.weber(at)vetmed.uni-giessen(dot)de
Projektbeschreibung
Viren sind aufgrund ihres relativ kleinen Genoms stark von Funktionen der Wirtszellen abhängig. Viele dieser Funktionen werden durch zellkodierte, post-translationale Proteinmodifikationen reguliert, gegen die eine beträchtliche Anzahl pharmazeutischer Inhibitoren verfügbar ist.
Rift Valley Fever Virus (RVFV) ist ein in Afrika endemischer, Mücken-übertragener Zoonose-Erreger. In einem typischen RVFV-Ausbruch sterben Tausende Nutztiere und Hunderte Menschen. In der vorherigen Förderperiode haben wir mittels einer high-throughput Screening Plattform einen pro-viralen Wirtszellfaktor für RVFV identifiziert, der an posttranslationale Proteinmodifikationen bindet. Inhibition dieses Faktors führte in human-Organoiden zu einer deutlichen Reduktion der viralen RNA-Synthese und der Virusproduktion. Zudem ergaben Proteom-Analysen, dass ein RVFV-Protein posttranslational so modifiziert wird, dass der neue Wirtsfaktor binden kann, und dass die Mutation dieser Modifikationsstelle die virale RNA-Synthese reduziert.
Wissenschaftliches Ziel:
Das geplante Projekt zielt darauf ab, essentielle Interaktionen von VP40 mit sich selbst durch synthetische Peptide oder kleine Moleküle zu hemmen und dadurch die Virusvermehrung zu blockieren. Eine der wesentlichen Herausforderungen dieses Projektes ist die Identifizierung von zellgängigen nicht (zyto)toxischen Inhibitoren.
DRUID-Kooperationspartner:
A1 Becker, A2 Grünweller, B2 Ziebuhr, C1 Bender/Hildt, D1 Friebertshäuser/Steinmetzer, E3 Rahlfs/ Przyborski, E4 Spengler, E6 Schiffmann, E7P Krijnse Locker
Literatur A3: 1. Wuerth & Weber (2016) Viruses 8, 174*. 2. Barr, Weber, Schmaljohn (2020) Fields Virology, vol 1, p 706-749*
A4Leitstruktursuche zur Inhibierung des Chaperons IpgC aus Shigella sp.
Andreas Heine, Klaus Reuter
Prof. Dr. Andreas Heine
Institut für Pharmazeutische Chemie
Marbacher Weg 6
35032 Marburg
Tel.: +49 (0)6421-28 21313
Fax: +49 (0)6421-28 28994
E-Mail: heinea(at)staff.uni-marburg(dot)de
Prof. Dr. Klaus Reuter
Institut für Pharmazeutische Chemie
Philipps-Universität Marburg
Marbacher Weg 6
35032 Marburg
Tel.: +49 (0)6421-28 25845
Fax: +49 (0)6421-28 28994
E-Mail: reuterk(at)staff.uni-marburg(dot)de
Projektbeschreibung
Bakterien der Gattung Shigella dringen in die Epithelzellen des Colons ein und verursachen eine entzündliche Erkrankung des Dickdarms. Letztere ist als „Bakterienruhr“ bekannt und fordert v.a. in Entwicklungsländern zahlreiche Todesopfer. Das Shigella-spezifische Chaperon IpgC ist für die Pathogenität des Erregers essentiell und tritt mit zahlreichen weiteren Pathogenitätsfaktoren in Wechselwirkung. In Abwesenheit eines „Substrat-Proteins“ liegt IpgC als Homodimer vor, was für die Aufrechterhaltung seiner Stabilität erforderlich ist. Wir nutzen IpgC als Zielprotein für die strukturbasierte Entwicklung von Wirkstoffen gegen Bakterienruhr, indem wir durch Kleinmoleküle die Homodimerbildung und/oder sein Binden an „Substrat-Proteine“ verhindern. Grundlage dafür ist ein von uns etabliertes Protokoll, welches reproduzierbar hervorragend streuende Kristalle von IpgC liefert. Ein bereits durchgeführtes Fragment-Screening führte zu mehreren Treffern, von denen einige mittlerweile zu größeren „Bindern“ erweitert werden konnten. Um deren Effekt auf die Homodimerbildung, auf die Wechselwirkung mit Substraten und auf die Stabilität des Proteins zu untersuchen, setzen wir neben der Proteinkristallographie Methoden wie „Mikroskalierte Thermophorese“, „Isothermische Titrationskalorimetrie“ sowie ein „Thermal Shift“-Assay ein.

Kristallstruktur des IpgC-Homodimers ©Klaus Reuter
An IpgC-gebundenes „erweitertes Fragment“ ©Marina Gardonyi
Wissenschaftliches Ziel:
Hauptziel ist neben zusätzlicher Strukturinformation über IpgC die Optimierung der bisher erhaltenen Verbindungen sowie ihr Einsatz als Leitstrukturen bei der Anti-Shigellose-Wirkstoffentwicklung.
DRUID-Kooperationspartner:
A1 Stephan Becker, B1 Wibke Diederich / Peter Kolb, B7 Franco Falcone, D1 Eva Friebertshäuser / Torsten Steinmetzer
Literatur A4: [1] Agerberth et al. (2005) World Health Organ [2] Williams & Berkley (2018) Paediatr Int Child Health 38:50-65. [3] Sansonetti (2001) Am J Physiol Liver Physiol 280:319-323. [4] Parsot et al. (2003) Curr Opin Microbiol 6:7-14. [5] Lunelli et al. (2009) Proc Natl Acad Sci USA 106:9661-9666.
A6Die hoch divergente Aktin-Superfamilie als Wirkstoffziel bei der Übertragung von Malariaparasiten
Ross Douglas
Dr. Ross Douglas
Biomedical Research Center Seltersberg (BFS)
Molecular Infections Biology
Justus Liebig Universität Giessen
Schubertstrasse 81
35392 Gießen
Tel.: +49 (0)641-99 39145
Fax: +49 (0)641-99 39129
E-Mail: ross.g.douglas(at)ernaehrung.uni-giessen(dot)de
Projektbeschreibung
Malaria ist nach wie vor eine der verheerendsten Krankheiten und wird durch einzellige Parasiten namens Plasmodium verursacht. Diese Parasiten werden durch Anopheles-Moskitos von Mensch zu Mensch übertragen. Der Parasit benötigt eine Reihe verschiedener Proteine, die ihm die Übertragung auf den Moskito-Vektor ermöglichen, darunter Mitglieder des stark divergierenden Aktin-Zytoskeletts und seiner Regulatoren. Das Zytoskelett des Parasiten hat einzigartige Eigenschaften, um die Übertragung zu ermöglichen. Da es für verschiedene Prozesse des Parasiten in unterschiedlichen Stadien des Lebenszyklus unerlässlich ist, enthält es vielversprechende Ziele für neuartige Malariatherapien. Wir nutzen Ansätze zur Target-Validierung, um neue Targets zu identifizieren und zu charakterisieren, die die Übertragung blockieren und zur Kontrolle der Krankheitsausbreitung eingesetzt werden könnten.
Der Ansatz der Target-Validierung hat neue Targets identifiziert, die die Übertragung blockieren.
Wissenschaftliches Ziel:
Unser Ziel ist es, die identifizierten Proteine mit in vitro und in vivo Methoden zu charakterisieren, um neue Wirkstoffe zu identifizieren, die selektiv auf diese Proteine abzielen und somit als Kandidaten für Medikamente zur Blockierung der Übertragung dienen.
DRUID-Kooperationspartner:
A7 Przyborski, B1 Diederich/Kolb, B7 P Falcone, E3 Rahlfs/Przyborski
Literatur A6: [1] Douglas et al. (2018) PLOS Bio e2005345; [2] Douglas et al. (2018) Malaria J 17:3191898-905; [3] Douglas et al. (2015) Trends Parasitol 31(8):357-362.
A7Plasmodium falciparum Chaperone, Co-Chaperone und deren Protein-Protein-Interaktionen als Ziele für neue Interventionsstrategien
Jude Przyborski
Prof. Dr. Jude Przyborski
Interdisziplinäres Forschungszentrum (iFZ)
Justus-Liebig-Universität Gießen
Heinrich-Buff-Ring 26-32
35392 Gießen
Tel.: +49 (0)641-99 39114
E-Mail: jude.przyborski(at)ernaehrung.uni-giessen(dot)de
Projektbeschreibung
Übersicht der Assay-Entwicklung
Malariaparasiten dringen in reife menschliche rote Blutkörperchen (RBC) ein und leben darin. Um sein Überleben zu sichern, baut der Parasit die von ihm gewählte Wirtszelle zu seinem eigenen Vorteil um. Infizierte rote Blutkörperchen werden klebrig und haften an der Innenwand kleiner Blutgefäße, außerdem überziehen sie sich mit Proteinen, die es ihnen ermöglichen, für das Immunsystem unsichtbar zu werden. Dies führt leider zur Krankheit des Patienten und schließlich zum Tod. Wir haben kürzlich eine Reihe wichtiger molekularer Akteure identifiziert, die für diesen Umbauprozess wesentlich sind, darunter Mitglieder der so genannten HSP70- und HSP40-Familien. Das Ziel dieses Projekts ist es, die Funktion von HSP40 und HSP70 zu blockieren. Wenn uns dies gelingt, werden die Parasiten wahrscheinlich aus dem Blutkreislauf entfernt, wodurch die Schwere der Krankheit gelindert wird. Zu diesem Zweck werden wir eine Reihe von Assays zur Messung der Aktivität von HSP40/HSP70 entwickeln und auf dieser Grundlage nach Verbindungen suchen, die diese Wechselwirkung verringern. Vielversprechende Verbindungen werden dann direkt an Parasiten auf ihre Fähigkeit getestet, die Veränderung der Wirtszellen zu verringern.
Literatur A7: 1. Diehl et al. (2021) PLoS Pathogens 17:e1009969 2. Zhang et al. (2017) Sci Rep 7: 42188 3. Charnaud et al. (2017) PLoS One 12: e0181656 4. Külzer et al. (2012) Cell Micro 14: 1784-95 5. Külzer et al. (2010) Cell Micro 12: 1398-1420
Projektbereich B:
B1Dengue- und Zikavirus, Design von Inhibitoren der NS3/NS2B-Serinprotease
Wibke Diederich, Peter Kolb
Prof. Dr. Wibke Diederich
Institut für Pharmazeutische Chemie
Zentrum für Tumor- und Immunbiologie
Philipps-Universität Marburg
Hans-Meerwein-Straße 3
35043 Marburg
Tel.: +49 (0)6421-28 25810
Fax: +49 (0)6421-28 26254
E-Mail: wibke.diederich(at)staff.uni-marburg(dot)de
Prof. Dr. Peter Kolb
Institut für Pharmazeutische Chemie
Philipps-Universität Marburg
Marbacher Weg 8
35032 Marburg
Tel.: +49 (0)6421-28 25908
Fax: +49 (0)6421-28 26652
E-Mail: peter.kolb(at)uni-marburg(dot)de
Projektbeschreibung
Infektionen mit dem Dengue-Virus haben in den letzten Jahren mit rund 50-100 Millionen Neuinfektionen pro Jahr einen neuen Höchststand erreicht. In der überwiegenden Zahl der Fälle verläuft die Infektion mit milden, grippeähnlichen Symptomen, jedoch entwickelt ein kleiner Prozentsatz der Betroffenen, häufig Kinder, ein hämorrhagisches Fieber, das bei schwerem Verlauf tödlich verläuft. Zwar steht inzwischen ein Impfstoff zur Verfügung, da dieser aber nur für einen sehr begrenzten Personenkreis zugelassen ist, ist die Entwicklung von Wirkstoffen, die die Vermehrung des Virus effizient unterdrücken, unabdingbar. Im Fokus unser Arbeiten steht die viruseigene Serinprotease NS3/NS2B, die das virale Vorläuferprotein in funktionelle Proteine spaltet und für Reifung des Virus unerlässlich ist.
Abb. B1. Kristallstruktur der NS3/NS2B Protease DENV 3.1
Wissenschaftliches Ziel:
Ziel des Projektes ist die Weiterentwicklung allosterischer Inhibitoren der viruseigenen Serinprotease NS3/NS2B, nicht nur hinsichtlich ihrer Affinität, sondern auch in Bezug auf ihre pharmakokinetischen Eigenschaften und ihre Toxizität (hit-to-lead-Entwicklung) mittels eines kombinierten Ansatzes aus computergestütztem Design, Synthese, biologischen Tests und der Kristallstrukturanalyse. Darüber hinaus sollen basierend auf den gewonnenen Erkenntnisse Inhibitoren der verwandten Zika-Protease entwickelt werden.
DRUID-Kooperationspartner:
A1 Becker, A2 Grünweller, B3 Rahlfs/Kolb/van Zandbergen, XY Herker, E6 Schiffmann/Laux
Literatur B1: [1] Noble et al. (2012) J Virol 86(1):438-446; *[2] Chevillard et al. (2015), J Chem Inf Model 55(9):1824-1835; *[3] Chevillard et al. (2018) J Med Chem, 61(3):1118-1129.
B2Virale Protein-de-ADP-Ribosylierungs- und NMPylierungs-Aktivitäten als potentielle therapeutische Angriffspunkte gegen Coronaviren
John Ziebuhr
Prof. Dr. John Ziebuhr
Institut für Medizinische Virologie
Biomedizinisches Forschungszentrum
Seltersberg (BFS)
Justus-Liebig-Universität Gießen
Schubertstraße 81
35392 Gießen
Tel.: +49 (0)641-99 41200
Fax: +49 (0)641-99 41209
E-Mail: John.Ziebuhr(at)viro.med.uni-giessen(dot)de
Projektbeschreibung
Coronaviren sind wichtige Pathogene von Mensch und Tier und verursachen v. a. respiratorische und intestinale Infektionen. Sie besitzen ein hohes zoonotisches Potential und haben wiederholt zu Ausbrüchen schwerer respiratorischer Infektionen beim Menschen geführt, wie die seit 2019 laufende SARS-CoV-2-Pandemie eindrücklich belegt. Die zur Verfügung stehenden therapeutischen Möglichkeiten für schwere Verlaufsformen von COVID-19 und anderen Coronavirusinfektionen sind extrem begrenzt, so dass die Entwicklung neuer Therapeutika eine hohe Priorität besitzt. Das Forschungsprojekt B2 konzentriert sich auf zwei coronavirale Proteine, die bei allen bekannten Coronaviren konserviert sind: die Makrodomäne (macD) im Nichtstrukturprotein 3 (nsp3) sowie eine neu entdeckte Nukleotidyltransferase (NiRAN), die mit der viralen RNA-Polymerase im nsp12 verbunden ist und für die Virusvermehrung essentiell ist.
Coronavirales Replikase-Polyprotein 1ab, aus dem durch proteolytische Spaltung (PL1, PL2, 3CL) 16 Nichtstrukturproteine mit zahlreichen enzymatischen und anderen Funktionen freigesetzt werden.
Wissenschaftliches Ziel:
Das Projekt zielt darauf ab, die biochemischen Eigenschaften der beiden Proteine umfassend zu charakterisieren und ihre Funktionen im viralen Replikationszyklus in geeigneten Zellkulturmodellen zu bestimmen. Auf Grundlage der konservierten Substratspezifität der NiRAN-Domäne sollen HTS-Assays entwickelt und zur Identifizierung potentieller Inhibitoren genutzt werden.
DRUID-Kooperationspartner:
A2 Grünweller, A3 Weber, C5 Kraiczy, E3 Rahlfs/Przyborski
Literatur B2: 1. *Slanina et al. (2021) Proc Natl Acad Sci USA 118: e2022310118 2. *Krichel et al. (2021) Sci. Adv. 7: eabf1004 3. *Müller et al. (2021) Antiviral Res 175: 1004706 4. *Snijder et al. (2016) Adv Virus Res 96: 59-126 5. *Putics et al. (2005) J Virol 79: 12721-31.
B3NAD(P)H-abhängige Stoffwechselwege als Ziele für neue Antiinfektiva
Stefan Rahlfs, Ger van Zandbergen, Peter Kolb
Dr. Stefan Rahlfs
Biochemie und Molekularbiologie
Justus-Liebig-Universität Gießen
Heinrich-Buff-Ring 26-32
35390 Gießen
Tel.: +49 (0)641-99 39117
E-Mail: stefan.rahlfs(at)ernaehrung.uni-giessen(dot)de
Prof. Dr. Ger van Zandbergen
Abteilung Immunologie
Paul-Ehrlich-Institut
Paul-Ehrlich-Str. 51-59
63225 Langen
Tel.: +49 (0)6103-77 2005
E-Mail: Ger.Zandbergen(at)pei(dot)de
Prof. Dr. Peter Kolb
Institut für Pharmazeutische Chemie
Philipps-Universität Marburg
Marbacher Weg 8
35032 Marburg
Tel.: +49 (0)6421-28 25908
Fax: +49 (0)6421-28 26652
E-Mail: peter.kolb(at)uni-marburg(dot)de
Projektbeschreibung
Das zelluläre Redoxgleichgewicht spielt für pathogene Mikroorganismen eine essentielle Rolle. Zentral daran beteiligt sind Enzyme des NAD(P)H-abhängigen Glutathion- und Thioredoxinsystems1,2, sowie die Glucose-6-Phosphatdehydrogenase (G6PD) und 6-Phosphogluconatdehydrogenase (6PGD), die maßgeblich zur Bereitstellung von NADPH und Ribose-5-Phosphat über den Pentosephosphatweg beitragen3. Die bifunktionale G6PD der Malariaparasiten Plasmodium falciparum und P. vivax (GluPho) unterscheidet sich funktionell und strukturell von den humanen Wirtsenzymen4 und ist essenziell für den Malariaparasiten5. In Kooperation mit dem Sanford-Burnham Institute/UCSD, La Jolla, konnten wir für die PfGluPho einen high-throughput kompatiblen Assay etablieren und ca. 400.000 Substanzen (u.a. NIH MLSMR Collection) screenen6. Anschließende Struktur-Wirkungs-Analysen und Lead-Optimierung resultierten in den im nanomolaren Bereich aktiven und hoch selektiven PfGluPho Inhibitor SBI-7507. Das Konzept konnte bereits auf Leishmania übertragen werden, die 3D-Kristallstruktur der Leishmania donovani G6PD und PGD gelöst werden und auf deren Basis ein erstes in silico Screening von Kleinmolekülen durchgeführt werden. Darüber hinaus wurde ein high-throughput kompatibler Assay für die rekombinanten G6PDs und 6PGDs aus Leishmania etabliert, um ein Inhibitor-Screening bei dem Novartis FAST lab in Cambridge (USA) durchzuführen.
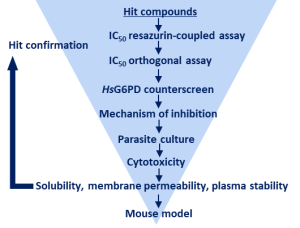
Workflow-Diagramm zur Inhibitor Identifizierung gegen G6PDs/PGDs
3D-Kristallstruktur LdG6PD ©Isabell Berneburg
Wissenschaftliches Ziel:
Ziel des Projektes ist die funktionelle und strukturelle Charakterisierung der Enzyme G6PD und 6PGD aus Leishmania und Plasmodium als Zielmoleküle für die Wirkstoffentwicklung, sowie die Identifizierung und Weiterentwicklung von Inhibitoren dieser Enzyme (in silico und HTS). Das Konzept soll auch auf weitere Erreger innerhalb des DRUID Konsortiums, wie z.B. Schistosoma, übertragen werden.
DRUID-Kooperationspartner:
B1 Diederich/Kolb; B4 Grevelding; D3 van Zandbergen; E1 Grevelding/Häberlein; E3 Rahlfs/Przyborski; E4 Spengler; E6 Schiffmann/Laux
Literatur B3: 1. Fritz-Wolf et al. (2011) Nature Comm. 2:383* 2. Koncarevic et al. (2009) PNAS 106: 13323-8* 3. Bozdech and Ginsburg (2005) Malaria J 3:23 4. Jortzik et al. (2011) Biochem J Energy 436:641-50* 5. Allen et al. (2015) FEBS J 282:3808-23*, 6. Preuss et al. (2012) J Med Chem 55:7262-72* 7. Berneburg et al. (2022) Antimicrob Agents Chemother (accepted)*
*eigene Publikationen
B4Schistosoma mansoni: Biologische Zielstrukturen und Inhibitorentwicklung
Christoph Grevelding
Prof. Dr. Christoph Grevelding
BFS, Institut für Parasitologie
Justus-Liebig-Universität Gießen
Schubertstraße 81
35392 Gießen
Tel.: +49 (0)641-99 38466
Fax: +49 (0)641-99 38469
E-Mail: Christoph.Grevelding(at)vetmed.uni-giessen(dot)de
Projektbeschreibung
Hintergrund: Beim dem human- und tierpathogenen Wurmparasiten Schistosoma mansoni hängt die sexuelle Reifung des Weibchens von einem kontinuierlichen Paarungskontakt mit dem Männchen ab (Abb. 1). Die Paarung ist somit auch für die Produktion von Wurmeiern und der daraus resultierenden Pathogenese der Schistosomiasis entschei-dend. Wir haben gezeigt, dass verschiedene Molekülklassen in diese Reproduktionsprozesse involviert sind.1 So offenbarten Versuche mit Kinaseinhibitoren und RNA-Interferenz gegen Kinasen, dass diese Moleküle Zellteilungsaktivität, Spermatogenese, Oogenese, Eiproduk-tion und Vitalität von Würmern in vitro negativ beeinflussen. Der Abl-TK-Inhibitor Imatinib (Krebsmedikament Glivec) induzierte in adulten S. mansoni eine gestörte Reproduktion sowie die Ablösung der Gastro-dermis mit letalen Folgen.2,3
Neben Kinasen, die wir in Kooperation mit der AG Falcone bearbeiten4, stehen weitere Enzyme im Fokus unserer Untersuchungen. Dazu zählen Aldehyddehydrogenasen (ALDH) und Aldehydreduktasen (AR), die durch molekularen Stress induzierte Prozesse in Schistosomen regulieren. Ihre Inhibition könnte den Parasiten schwächen. Erste in vitro-Experimente mit Inhibitoren gegen diese Moleküle, wie z.B. dem ALDH-Inhibitor Disulfiram, führten in kurzer Zeit zu morphologischen Veränderungen, Entpaarung, reduzierter Vitalität sowie zum Tod des Parasiten in vitro. Aber auch Helikasen können interessante Angriffspunkte darstellen, wie Arbeiten mit dem RNA-Helikase-Inhibitor Silvestrol gezeigt haben.5 In adulten S. mansoni reduzierte Silvestrol die Fitness adulter Würmer in vitro und die Stammzellproliferation in Keimzellen (Abb. 2). Mit den AGs Prof. Schlitzer und Prof. Grünweller (Marburg) werden wir an diesen Zielmolekülen arbeiten und an der Entwicklung synthetischer Inhibitoren6,7, um physiologische und morphologische Effekte in vitro und bei Topkandidaten dann im Infektionsmodell in vivo zu untersuchen.

Abb. 1. Lichtmikroskopische Aufnahme eines Schistosomen-Pärchens in vitro. Während des dauerhaften Paarungskontaktes liegt das Weibchen (Pfeil) in einer Bauchfalte des Männ-chens. Das ist eine wesentliche Voraussetzung für die Eiproduk-tion (Sterne).
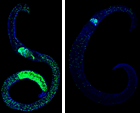
Abb. 2. EdU-Färbung von S. mansoni-Weibchen vor (links) und nach Silvestrol-Behandlung (rechts). Die Anzahl der Stammzellen (grün) ist signifikant redu-ziert (rechts).
Wissenschaftliches Ziel:
Wir werden die Klonierung sowie die molekulare und biochemische Charakterisierungen zweier RNA-Helikasen aus S. mansoni durchführen. Unterstützt durch verschiedene Kooperationspartner/innen im DRUID-Konsortium (s.u.) werden wir die rekombinanten Proteine in Enzymassays gegen Silvestrol und synthetische Derivate testen. Ferner planen wir, mit den bereits rekombinant hergestellten Enzymen SmALDH312 und SmAR, weitere potenzielle Zielmoleküle, Enzymtests mit Inhibitoren durchzuführen. Hierzu zählen u.a. Disulfiram-derivate, die bereits anti-schistosomale Effekte in vitro gezeigt hatten. Experimenten zur Kristallisationen und Strukturaufklärungen dieser Moleküle werden auch durchgeführt. Ziel ist die Entwicklung von Wirkstoffen mit inhibitorischer Aktivität gegen die ausgewählten Zielproteine, hoher Spezifität und Bioverfügbarkeit bei gleichzeitig reduzierter Toxizität.8 In vitro– und später auch in vivo-Experimente sollen die Übertragbarkeit der gewonnenen Erkenntnisse auch auf andere Parasiten zeigen (Plattformprojekt E1).
DRUID-Kooperationspartner:
A2 AG Grünweller, B3 AG Rahlfs/Kolb/van Zandbergen, B5 AG Schlitzer, E1 Platformprojekt/Häberlein, E3 AG Rahlfs/Przyborski, E5 AG Czermak/Salzig lab
Literatur B4: 1Beckmann et al. (2010) PLoS Pathog 6:e1000769; 2Beckmann et al. (2010) Int J Parasitol 40:521-6; 3Beckmann et al. (2012) Curr Pharm Des 18:3579-94; 4Moreira et al. (2022) Molecules (in press); 5Taroncher-Oldenburg et al. (2021) Microorganisms 9(3):540; 6Peter Ventura et al. (2021) ChemMedChem 14(21):1856-62; 7Peter Ventura et al. (2012) Arch Pharm (Weinheim) 354(12):
B5Entwicklung von Dithiocarbamaten als Anthelminthika und Hemmstoffen der RNA-Helikase eIF4A als potentielle antivirale Wirkstoffe
Martin Schlitzer
Prof. Dr. Martin Schlitzer
Institut für Pharmazeutische Chemie
Fachbereich Pharmazie
Philipps-Universität Marburg
Marbacher Weg 6
35037 Marburg
Tel.: +49 (0)6421 28-25840
E-Mail: schlitzer(at)staff.uni-marburg(dot)de
Projektbeschreibung
Wurmerkrankungen stellen ein großes Problem in großen Teilen der Welt dar. Verglichen mit der Krankheitslast ist die Zahl der zur Verfügung stehenden Wirkstoffe gering. In zahlreichen aufeinanderfolgenden Zyklen von Design, Synthese und Testung soll die neue Wirkstoffklasse der Dithiocarbamate hinsichtlich ihrer Wirkung an verschiedenen Helminthen, ihrer Wirtstoxizität und ihrer generellen Wirkstoffeigenschaften optimiert werden. Derivate werden synthetisiert, auf ihre Wirkung auf verschiedene pathogene Würmer und ihre Toxizität gegen humane Zellen getestet. Von vielversprechenden Derivaten werden ADME-Parameter bestimmt und einzelne Verbindungen ggf. einer in-vivo Testung zugeführt.
Diverse humanpathogen Viren, u.a. auch das Ebola- und das SARS-CoV-2-Virus, sind für ihre intrazelluläre Vermehrung auf Wirtsfaktoren angewiesen, die damit geeignete Zielstrukturen für die Entwicklung anti-viraler Wirkstoffe darstellen. Einer dieser Wirtsfaktoren ist die RNA-Helicase eIF4A. Basierend auf der Kristallstruktur werden potentielle Liganden konstruiert, in den Protein-RNA-Komplex gedockt und bei guter Passung synthetisch hergestellt. Die Verbindungen werden auf ihre Wirkung hinsichtlich der Translationseffizienz getestet. Gut wirksame Derivate werden anschließend auf ihren Hemmeffekt auf die Virusvermehrung in Zellkulturen getestet.

Wissenschaftliches Ziel:
Ziel in beiden Projekten ist es, Wirkstoffmoleküle zu entwickeln, die die Qualität einer Leitstruktur oder sogar eines Entwicklungskandidaten aufweisen.
DRUID-Kooperationspartner:
A2, A3, A4, A7, B2, B7P, C6 NWG, D4, E1, E4, E6
Literatur B5: –
B6 PIdentifizierung von antiviralen Targets im Lipidmetabolismus
Eva Herker
Prof. Dr. Eva Herker
Institut für Virologie
Philipps-Universität Marburg
Hans-Meerwein-Str. 2
35043 Marburg
Tel.: +49 (0)6421-28-64525
E-Mail: eva.herker(at)uni-marburg(dot)de
Projektbeschreibung
Die humanpathogenen Flaviviren Dengue (DENV), Gelbfieber (YFV), Zika (ZIKV), West-Nil (WNV) und das Frühsommer-Meningoenzephalitisvirus (TBEV/FSME) verursachen akute Infektionen mit teils schweren Komplikationen. Fundamentale Schritte in der Flavivirusreplikation sind eng mit zellulären Lipiden verknüpft. Hierzu zählen unter anderem Membranreorganisationen zur Bildung der Replikationsvesikel oder die Bildung der Virushülle. Flaviviren verändern die Lipidkomposition der Wirtszelle zu ihrem Vorteil, und die Aktivität verschiedener lipidmetabolisierender Enzyme ist essentiell für die erfolgreiche Replikation. Aus diesem Grund stellen Enzyme verschiedener Lipidstoffwechselwege interessante Targets für eine (pan-) antiflavivirale Therapie dar. Allerdings ist noch nicht detailliert geklärt, ob die o.g. Vertreter der Flaviviren auf unterschiedliche oder ähnliche Zweige des Lipidmetabolismus angewiesen sind.
Immunfluoreszenzmikroskopie von Zellen, die mit verschiedenen Flaviviren infiziert sind; rot: virales E-Protein; grün: lipid droplet
Wissenschaftliches Ziel:
Das Projekt zielt darauf ab, antivirale Angriffspunkte im zellulären Lipidmetabolismus zu identifizieren. Hierbei wird die Rolle verschiedener Schlüsselenzyme der de novo Fettsäure- und Cholesterol-Biosynthese, des Phospholipid- und Neutrallipidstoffwechsels, sowie von Enzymen der Lipidremodellierung in der Flavivirusreplikation mittels eines shRNA-basierten Screenings analysiert. Zusätzlich werden verschiedene Inhibitoren getestet. Die Ergebnisse werden in verschiedenen Zelltypen validiert und die molekularen Mechanismen detailliert untersucht.
DRUID-Kooperationspartner:
B1 Diederich/Kolb, D1 Böttcher-Friebertshäuser/Steinmetzer, E7P Krijnse-Locker, E4 Spengler
Literatur B6P: 1. Herker et al. (2010) Nat Med 16: 1295 2. Harris et al. (2011) J Biol Chem 286: 42615 3. Herker et al. (2012) J Biol Chem 287: 2280 4. Rosch et al. (2016) Cell Rep 16: 3219 5. Hofmann et al. (2018) Biochim Biophys Acta Mol Cell Biol Lipids 1863: 1041 6. Schobel et al. (2018) Sci Rep 8: 3893 7. Lassen et al. (2019) J Cell Sci 132: jcs.217042 8. Bley et al. (2020) Int J Mol Sci 21: 9. Herker et al. (2021) Trends Cell Biol 31: 345 10. Nguyen-Dinh et al. (2021) Cells 10: 2407
B7PNeue Wege zur Kontrolle von Schistosomiasis und Echinokokkose
Franco Falcone
Prof. Dr. Franco Falcone
Institut für Parasitologie
BFS - Biomedizinisches Forschungszentrum Seltersberg
Justus-Liebig-Universität Gießen
Schubertstraße 81
35392 Gießen
Tel.: +49 (0)641-99 38030
E-Mail: franco.falcone(at)vetmed.uni-giessen(dot)de
Projektbeschreibung
Echinokokkose und Schistosomiasis sind zwei sogenannte vernachlässigte Tropenkrankheiten, die die Gesundheit der Betroffenen in endemischen Ländern stark beeinträchtigen. Unsere Gruppe entwickelt neue Instrumente, die für eine bessere Kontrolle dieser beiden Krankheiten eingesetzt werden können. Für Bilharziose entwickeln wir neue Medikamente, die auf die schistosomalen Mitogen-aktivierten Proteinkinasen abzielen [1], um neue Verbindungen zu finden, die sowohl larvizid als auch adultizid sind. Für die Echinokokkose entwickeln wir eine neuartige Diagnoseplattform, die auf dem Nachweis von parasitenspezifischem IgE unter Verwendung von humanisierten IgE-Reporterzelllinien beruht [2-4], die in verschiedenen Formaten eingesetzt werden können. Wir arbeiten auch an der Ausweitung dieser Diagnoseplattform auf andere parasitäre Infektionen (z. B. Fasciola, Zystizerkose, Clonorchis, Opisthorchis).
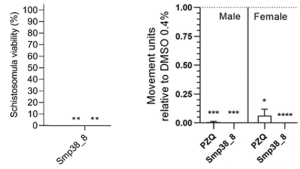
Larvizide und Adultizide Aktivität von Verbindung 38_8, die aus einer Substanzbibliothek mittels unseres bionformatischen Ansatzes ausgesucht wurde.
Strukturvorhersage von S. mansoni JNK im Vergleich zum humanen Orthologen
Wissenschaftliches Ziel:
Das Schistosomen-Projekt zielt darauf ab, mithilfe einer bioinformatischen in silico Analyse Leitmoleküle für Kinase-Inhibitoren zu identifizieren und diese zu geeigneten Arzneimitteln weiterzuentwickeln. Im Rahmen des Echinococcus-Projekts streben wir ein besseres Verständnis der schützenden Immunreaktionen gegen Echinokokken an und wollen dieses Wissen in die Entwicklung von Impfstoffen und stark verbesserten, innovativen Diagnosetechnologien einfließen lassen.
DRUID-Kooperationspartner:
A4 Heine lab, B4 Grevelding lab, C6 NWG Häberlein lab, E1 Czermak/Salzig lab
Literatur B7P: [1] *Pereira-Moreira et al. (2020) ACS Omega 5:9064-9070 [2] Kalli M et al. (2020) Sci Rep. 10:18208 [3] Kalli M. et al. (2020) Methods Mol Biol.;2163:155-162. [4] Prakash PS et al. (2021) Parasitol Res.
Projektbereich C:
C1Targets für antivirale Strategien gegen das Zika Virus
Daniela Bender, Eberhard Hildt
Dr. Daniela Bender
Paul-Ehrlich-Institut
Bundesinstitut für Impfstoffe
und biomedizinische Arzneimittel
Paul-Ehrlich-Straße 51-59
63225 Langen
Tel.: +49 (0)6103-77 5411
E-Mail: Daniela.Bender(at)pei(dot)de
Prof. Dr. Eberhard Hildt
Bundesinstitut für Impfstoffe
und biomedizinische Arzneimittel
Paul-Ehrlich-Institut
Paul-Ehrlich-Straße 51-59
63225 Langen
Tel.: +49 (0)6103-77 2140
Fax: +49 (0)6103-77 1234
E-Mail: Eberhard.Hildt(at)pei(dot)de
Projektbeschreibung
Zika-Viren (ZIKV) sind Arboviren aus der Familie der Flaviviren. Im Zuge der ZIKV Epidemie 2015/2016 hat die WHO am 01.02.2016 den Ausbruch in Südamerika zum Public Health Emergency of International Concern (PHEIC) erklärt, da es einen Zusammenhang zwischen der Zika-Virus-Infektion Schwangerer und dem Auftreten von Mikrozephalien bei Neugeborenen gibt. Derzeit steht weder eine Impfung noch eine spez. Therapie zur Verfügung. Durch Hemmung der Virusreplikation bereits in der frühen Phase der Infektion ggf. mittels einer temporären präventiven Strategie könnte die Viruslast deutlich reduziert und so die Verbreitung des Virus und so auch das Risiko einer intrauterinen Infektion vermindert werden. Im Rahmen der ersten Förderperiode konnten Zielstrukturen für antivirale Ansätze identifiziert werden.
Intrazelluäre Verteilung von Tetherin (rot) und dem ZIKV Hüllprotein E (grün) in Zellen infiziert mit zwei verschiedenen ZIKV Isolaten (Uganda bzw. Polynesia)
Wissenschaftliches Ziel:
Basierend auf bereits identifizierten Zielstrukturen und weiterer Zielstrukturen sollen antivirale Strategien entwickelt, der Mechanismus charakterisiert und deren Wirkung auch auf weitere Flaviviren untersucht werden.
DRUID-Kooperationspartner:
A2 AG Grünweller, B1 AG Diderich/Kolb, B6P AG Herker, C2 AG Kempf, C5 AG Glebe/Geyer, D1 AG Steinmetzer, E6 AG Schiffmann/Laux
Literatur C1: 1. Herrlein et al. (2021) J Virol. doi: 10.1128/jvi.02117-2 2. Sabino et al (2021)., J Virol. doi: 10.1128 3. *Maddaluno et al., 2020 EMBO Mol Med. doi: 10.15252/emmm.201911793 4. Basic et al. 2019 Antiviral Res. doi: 10.1016/j.antiviral.2019.104644. 5. Akhras et al. (2019) Viruses doi: 10.3390/v11080748. 6. Sabino et al., (2019) doi: 10.3390/v11060524 7. Elgner et al (2018) Viruses doi: 10.3390/v10040149
C2Bartonella bacilliformis Pathogenitätsfaktoren als diagnostische und therapeutische Targets
Volkhard Kempf
Prof. Dr. Volkhard A. J. Kempf
Universitätsklinikum Frankfurt
Institut für Medizinische Mikrobiologie
und Krankenhaushygiene
Goethe-Universität Frankfurt/Main
Paul-Ehrlich-Str. 40
60596 Frankfurt/Main
Tel.: +49 (0)69-6301 5019
Fax: +49 (0)69-6301 83431
E-Mail: volkhard.kempf(at)kgu(dot)de
Projektbeschreibung
Bartonella bacilliformis ist der Erreger der Carrión-Krankheit, eine in den Anden Südamerikas endemisch vorkommende und vektorübertragene Infektionserkrankung. Infektionen mit B. bacilliformis führen zu einem schweren hämolytischen Fieber mit hoher Letalität. Die Inhibition der bakteriell vermittelten Hämolyse stellt einen vielversprechenden Therapieansatz dar. Zwei Pathogenitätsfaktoren spielen bei der Hämolyse eine zentrale Rolle, von denen mindestens eines ein potentielles drug target darstellt. Bislang ist der Mensch der einzig bekannte Reservoir-Wirt für B. bacilliformis und es wird angenommen, dass asymptomatisch Infizierte die Quelle neuer Ausbrüche sind. Die Identifizierung asymptomatischer Träger ist daher von besonderer Bedeutung für die Infektionskontrolle. Hierfür wurde eine Alphaversion eines B. bacilliformis IgG ELISAs und Lineblots entwickelt.

Rasterelektronen-mikroskopie eines, durch B. bacilliformis, infizierten Erythrozyten.
B. bacilliformis NovaLisa® KIT.
Wissenschaftliches Ziel:
Ziel des Projektes ist es, die beiden Pathogenitätsfaktoren in Hinblick auf Hämolyse zu untersuchen, um neue Antivirulenzstrategien zu entwickeln. Die Alphaversion des B. bacilliformis IgG ELISAs und Lineblots soll, in Kooperation mit unseren Partnern aus Lima/Peru, in Feldstudien evaluiert werden.
DRUID-Kooperationspartner:
A7 Przyborski, C1 Bender / Hildt, E7 Locker, Gold Standard Diagnostics (früher NovaTec)
Literatur C2: [1] Garcia-Quintanilla et al. (2019) Parasites&Vectors 12(1):141, [2] Riess et al. (2004) J Exp Med 200:1267-78, [3] Dichter et al. (2019) Microbiology Res Announc, DOI: 10.1128/MRA.01377-19, [4] Dichter et al. (2021) Lancet Microbe 2:e685–94.
C3Komplement-interagierende Proteine von Rückfallfieberborrelien
Peter Kraiczy
Prof. Dr. Peter Kraiczy
Goethe-Universität Frankfurt/Main
Institut für Medizinische Mikrobiologie und Krankenhaushygiene
Universitätsklinikum Frankfurt
Paul-Ehrlich-Str. 40
60596 Frankfurt am Main
Tel.: +49 (0)69-6301 7165
Fax: +49 (0)69-6301 83431
E-Mail kraiczy(at)em.uni-frankfurt(dot)de
Projektbeschreibung
Borrelia recurrentis zählt zu den „neglected arthropod-borne pathogens“ und ist der Erreger des epidemischen Läuserückfallfiebers, das unbehandelt eine Mortalität von bis zu 40% aufweist. Die hämatogene Ausbreitung des Erregers lässt auf effiziente Strategien der Immunevasion schließen, um insbesondere dem Komplementsystem, zu entgehen. Dabei spielen Komplement-interagierende Proteine eine bedeutende Rolle. Mit Hilfe von bioinformatischen Analysen wurde ein Cluster von fünf Proteinen identifiziert, die auf unterschiedlichen Aktivierungsstufen das Komplementsystem inhibieren. Diese Proteine stellen vielversprechende Kandidaten für die Entwicklung von in vitro-Diagnostika dar. Zwei Testsysteme (Line immunoblot und ELISA) zur Diagnose des epidemischen Läuserückfallfiebers wurden bereits entwickelt und evaluiert.
Immunfluoreszenz-mikroskopische Aufnahme von B. recurrentis.
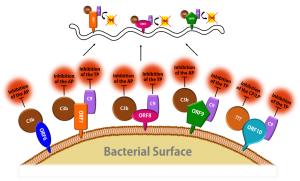
Schematische Darstellung der Komplementinaktivierung auf der Oberfläche von B. recurrentis.
Wissenschaftliches Ziel:
Der Fokus dieses Projektes liegt auf der funktionellen und strukturellen Charakterisierung weiterer Komplement-inhibierender Proteine von Borrelia recurrentis sowie der Optimierung der bestehenden Testsysteme und der Entwicklung eines point-of-care Antigen-Schnelltests.
DRUID-Kooperationspartner:
B2 Ziebuhr lab, C2 Kempf lab, C4 Steinhoff lab, D1 Steinmetzer lab, E3 Rahlfs/Przyborski, Gold Standard Diagnostics (früher NovaTec)
Literatur C3: [1] Cordes et al. (2005) Nat Struct Mol Biol 12:276-277; [2] Röttgerding et al. (2017) Sci Rep 7:303; [3] Nguyen et al. (2018) Front Cell Infect Microbiol. 8:23; [4] Walter et al. (2019) Front Immunol 10:2722; [5] Röttgerding and Kraiczy (2020) Front Immunol 11:1560; [6] Schmidt et al. (2021) Sci Rep 11:4964.
C4Verbesserte Diagnose und Behandlung der viszeralen Leishmaniose
Ulrich Steinhoff
Prof. Dr. Ulrich Steinhoff
Institut für Medizinische Mikrobiologie und Krankenhaushygiene
Philipps-Universität Marburg
Hans-Meerwein-Straße 2
35043 Marburg
Tel.: +49 (0)6421-28 66134
Fax: +49 (0)6421-58 66420
E-Mail: ulrich.steinhoff(at)staff.uni-marburg(dot)de
Projektbeschreibung
Die zuverlässige Diagnose sowie Behandlung der an viszeraler Leishmaniose (VL) erkrank-ten Menschen und Hunden (Reservoirwirt) ist für die Kontrolle dieser Infektion wichtig. Derzeit verfügbare diagnostische Tests, die auf der Antikörperreaktion mit dem Kinesin-protein von Leishmanien basieren, zeigen in manchen Endemiegebieten eine schlechte Sensitivität. Wir haben ein Kinesinantigen (rKLi8.3) entwickelt und patentiert, dass bei Mensch und Tier eine verbesserte diagnostische Leistung aufweist. Dies war möglich, indem wir ein Kinesinantigen mit optimierter Struktur (Repeats) und Sequenz entwickelt haben. Derzeit werden verschiedene Testformate mit rKLi 8.3 hergestellt und geprüft.
Die Behandlung der VL ist hinsichtlich von Wirksamkeit und Nebenwirkungen sehr proble-matisch. Es wurde ein Inhibitor (GNF-6702) der selektiv das Proteasom der Kinetoplastida (T. brucei, T. cruzei und L. donovani) inhibiert entwickelt. GNF-6702 bindet an eine Proteasomenuntereinheit von Kinetoplastida, die sich vom Menschen strukturell unter-scheidet. Die spezifische Bindung an das Kinetoplastenproteasom scheint ursächlich für die sehr geringe Toxizität in Säugerzellen zu sein. Wir konnten zeigen, dass die proteaso-male beta 4 Untereinheit bei allen bisher getesteten Leishmanienisolaten konserviert ist und sich somit als therapeutisches Ziel eignet.
Wissenschaftliches Ziel:
Im Projektteil Diagnose produzieren und überprüfen wir in Zusammenarbeit mit unserem Industriepartner verschiedene Formate eines sero-diagnostischen VL-Schnelltest. Im Projektteil Behandlung validieren wir in Zellkultur-experimenten Spezifität, Wirkung und Toxizität neuer, Kinetoplastida-spezifischen Proteasomen Inhibitoren.
DRUID-Kooperationspartner:
B1 Diederich/Kolb Labor, C2 Kempf Labor, D3 van Zandbergen Labor
Literatur C4: [1] Abass et al. (2013) PLoS Negl Trop Dis. 18;7; [2] Abass et al. (2015) PLoS One. 3;10; [3] Martínez Abad et al. (2017) Acta Trop. 166:133-138; [4] Pereira et al. (2020) Eur J Microbiol Immunol 27;10:165-171. [5] Khare et al. (2016) Nature 537: 229-233
C5Hit-to-Lead Entwicklung selektiver HBV/HDV Entry-Inhibitoren
Joachim Geyer, Dieter Glebe
Prof. Dr. Joachim Geyer
Institut für Pharmakologie und Toxikologie
Justus-Liebig-Universität Gießen
Schubertstraße 81
35392 Gießen
Phone: +49 (0)641-99 38404
E-Mail: joachim.m.geyer(at)vetmed.uni-giessen(dot)de
Prof. Dr. Dieter Glebe
Institut für Medizinische Virologie
Justus-Liebig-Universität Gießen
Schubertstraße 81
35392 Gießen
Phone: +49 (0)641-99 41246
E-Mail: dieter.glebe(at)viro.med.uni-giessen(dot)de
Projektbeschreibung
Infektionen mit den Hepatitis B (HBV) und D (HDV) Viren sind die Hauptursache für hepatozelluläres Karzinom und Leberzirrhose infolge chronischer Hepatitis. Obwohl ein wirksamer prophylaktischer Impfstoff zur Verfügung steht sind die therapeutischen Möglichkeiten insbesondere für HDV beschränkt. Die Entwicklung von HBV/HDV Entry-Inhibitoren könnte hier Abhilfe schaffen. Diese sollten spezifisch den HBV/HDV Rezeptor NTCP (einen physiologischen Gallensäuretransporter in der Leber) blockieren. Bisher wurden mehr als 200 Verbindungen aus verschiedenen Substanzklassen getestet und entsprechende 3D-Struktur-Wirkungsmodelle (QSAR) generiert. Des Weiteren wurde ein Pharmakophor-Modell für HBV/HDV Entry-Inhibitoren etabliert. Mit diesen Modellen wurden bereits virtuelle Substanzbibliotheken durchmustert und dabei weitere wirksame Hits identifiziert.
NTCP ist ein physiologischer Gallensäuretransporter in der Plasmamembrane von Leberzellen. NTCP ist aber auch der hepatische Rezeptor für HBV und HDV. HBV/HDV Entry-Inhibitoren sollten selektiv die Virusrezeptorfunktion von NTCP blockieren. 3D-QSAR- und Pharmacophor-Modelle helfen neue wirksame Verbindungen zu identifizieren.
Wissenschaftliches Ziel:
Entwicklung niedermolekularer oral bioverfügbarer HBV/HDV Entry-Inhibitoren, welche in der Lage sind selektiv die Virusrezeptorfunktion von NTCP zu blockieren, ohne dabei dessen physiologische Gallensäuretransportfunktion zu beeinträchtigen. Erste Hits sollen mittels molekularem Wirkstoffdesign zu Leitstrukturen weiterentwickelt werden.
DRUID-Kooperationspartner:
B1 Diederich/Kolb, B3 Rahlfs/Kolb/van Zandbergen, D2 Pfeiffer/Zeuzem/Hildt, E3 Rahlfs/Przyborski, E6 Schiffmann/Laux
Literatur C5: 1. *Kirstgen et al. (2020) Sci Rep 10:21772 2. *Grosser et al. (2021) Front Mol Biosci 8:689757 3. *Kirstgen et al. (2021) Viruses 13:666 4. *Kirstgen et al. (2021) Viruses 13:1489. 5. Paraskevopoulou et al. (2020) Proc Natl Acad Sci USA 117(30).
C6Das Fasciola-Kinom als Quelle für neue Wirkstofftargets
Simone Häberlein
PD Dr. Simone Häberlein
Justus-Liebig-Universität Gießen
BFS, Institut für Parasitologie
Schubertstraße 81
35392 Gießen
Tel.: +49 (0)641-99 38476
Fax: +49 (0)641-99 38469
E-Mail: Simone.Haeberlein(at)vetmed.uni-giessen(dot)de
Projektbeschreibung
Proteinkinasen regulieren eine Vielzahl zellulärer Prozesse und stellen vielversprechende Ziele für Medikamente dar, nicht nur in der Krebstherapie sondern auch gegen parasitäre Infektionserreger. Beide Erkrankungen haben u.a. gemein, dass Stammzellen eine besondere Rolle beim Wachstum spielen. Unsere Hypothese ist, dass die Inhibierung ausgewählter Proteinkinasen als Therapieansatz gegen den Leberegel Fasciola hepatica genutzt werden kann, einem weltweit vorkommenden Zoonose- und NTD-Erreger. Im Rahmen des Projektes werden (1) potentielle Wirkstoffziele im Fasciola-Kinom bioinformatisch identifiziert und genetisch validiert, (2) gegen den Leberegel aktive Kinase-Inhibitoren identifiziert, sowie (3) die Wirkungsweise von Kinase-Inhibitoren mit biochemischen und bildgebenden Methoden charakterisiert.
U.a. haben wir in Kooperation mit der AG Spengler (Projekt E4) den Einsatz der bildgebenden AP-MALDI-Massenspektrometrie für “Drug Imaging” in Parasiten-gewebe etabliert und können so Aufnahmeroute, -kinetik und Wirkstofftropismus von Kinase-inhibitoren in Leberegeln unter-suchen.
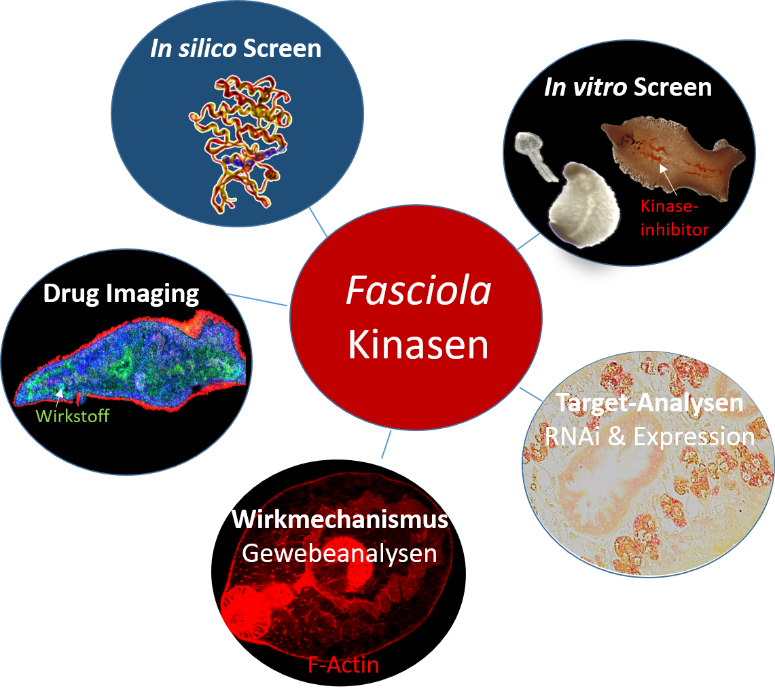
Strategie zur Identifizierung von Proteinkinase-Inhibitoren als Wirkstoffkandidat gegen den Leberegel Fasciola hepatica. ©Simone Häberlein
Wissenschaftliches Ziel:
Ziel des Projekts ist es, Proteinkinase-Inhibitoren als neue Wirkstoff-kandidaten gegen Fasciolose zu finden.
DRUID-Kooperationspartner:
A2 Grünweller lab, B4 Schlitzer lab, B5 Grevelding lab, B7 Falcone lab, E4 Spengler lab
Literatur C6: 1. Houhou et al. (2019) Sci Rep 9:15867. 2. Li et al. (2019) Parasitol Res 118(3):881-890. 3. Morawietz et al. (2020) Front Vet Sci 7:611270. 4. Mokosch et al. Anal Bioanal Chem 413(10): 2755-2766. 5. Morawietz et al. (2022) Parasitol Res (online ahead of print) doi: 10.1007/s00436-021-07388-1
Projektbereich D:
D1Hemmung virusaktivierender Wirtsproteasen
Eva Friebertshäuser, Torsten Steinmetzer
Prof. Dr. Eva Friebertshäuser
Institut für Virologie
Philipps-Universität Marburg
Hans-Meerwein-Str. 2
35043 Marburg
Tel.: +49 (0)6421-28 66019
Fax: +49 (0)6421-28 68962
E-Mail: friebertshaeuser(at)staff.uni-marburg(dot)de
Prof. Dr. Thorsten Steinmetzer
Institut für Pharmazeutische Chemie
Philipps-Universität Marburg
Marbacher Weg 10
35032 Marburg
Tel.: +49 (0)6421-28 25900
Fax: +49 (0)6421-28 25901
E-Mail: torsten.steinmetzer(at)staff.uni-marburg(dot)de
Projektbeschreibung
Die Reifespaltung viraler Hüllproteine durch Wirtsproteasen ist essentiell für die Infektiosität vieler humanpathogener Viren. Zahlreiche Oberflächenglykoproteine, u.a. hochpathogener aviärer Influenzaviren (z.B. H5N1), des Chikungunyavirus oder von Dengue-, West-Nil und Zika-Virus, werden durch furinartige Serinproteasen aktiviert. Andere Virusproteine, wie das Hämagglutinin zoonotischer H7N9 und saisonaler Influenza-A-Viren oder das Spike-Protein S vieler Coronaviren (CoV) werden durch die membranständige trypsinartige Serinprotease TMPRSS2 gespalten. Zuletzt konnten wir zeigen, dass das SARS-CoV-2 S sowohl durch Furin als auch TMPRSS2 aktiviert wird. Daher sind diese Wirtsproteasen vielversprechende Targets zur Entwicklung neuartiger antiviraler Wirkstoffe mit breitem Wirkungsspektrum.
Kristallstruktur des Furins im Komplex mit Inhibitor MI-1851.

Kristallstruktur der TMPRSS2 überlagert mit den Inhibitoren MI-1904 (gelbe) und MI-432 (orange).
Wissenschaftliches Ziel:
Im Rahmen des Projekts sollen strukturbasiert hochwirksame Hemmstoffe der virusaktivierenden Wirtsproteasen Furin und TMPRSS2 entwickelt, charakterisiert und auf ihre antivirale Wirksamkeit in Zellkultur und im Tiermodell geprüft werden.
DRUID-Kooperationspartner:
A1 Becker lab, A2 Grünweller lab, B6 Herker lab, C1 Hildt lab
Literatur D1: 1. Böttcher et al. (2006) J Virol 80: 9896-8 3. Becker et al. (2012) J Biol Chem 287: 21992-03 4. Böttcher-Friebertshäuser et al. (2012) Vaccine 30: 7374-80 5. Ivanova et al. (2017) ChemMedChem 12: 1953-68 6. Lam van et al. (2019) ChemMedChem 14, 673-85 7. Bestle et al. (2020) LSA 3: e202000786 8. Bestle et al. (2021) J Virol 95: e0090621 9. Lam van et al. (2021) ACS Med Chem Lett 12: 426-32.
D2HEV, Identifizierung zellulärer Targets für antivirale Strategien – Hemmung der Virusfreisetzung durch Modulation des Cholesterolspiegels
Kai-Henrik Peiffer, Stefan Zeuzem, Eberhard Hildt
Dr. Kai-Henrik Peiffer
Goethe-Universität Frankfurt am Main
Universitätsklinikum
Zentrum der Inneren Medizin
Medizinische Klinik I
Theodor-Stern-Kai 7
60596 Frankfurt am Main
E-Mail: kai-henrik.peiffer(at)kgu(dot)de
Prof. Dr. Eberhard Hildt
Bundesinstitut für Impfstoffe
und biomedizinische Arzneimittel
Paul-Ehrlich-Institut
Paul-Ehrlich-Straße 51-59
63225 Langen
Tel.: +49 (0)6103-77 2140
Fax: +49 (0)6103-77 1234
E-Mail: Eberhard.Hildt(at)pei(dot)de
Prof. Dr. Stefan Zeuzem
Department of Medicine
Goethe University Hospital
Theodor-Stern-Kai 7
60590 Frankfurt am Main
Tel.: +49 (0)69-6301 4544
E-Mail: zeuzem(at)em.uni-frankfurt(dot)de
Projektbeschreibung
Das Hepatitis E Virus führt jährlich zu über 20 Mio. Infektionen weltweit und betrifft Industrienationen, besonders allerdings Entwicklungsländer. Limitierte Behandlungs-optionen sind häufig mit schweren Nebenwirkungen verbunden. Da das unbehüllte Virus für seine Freisetzung endosomale Prozesse nutzt, bietet dies einen vielversprechenden Ansatzpunkt für neue Wirkstoffe. Im Rahmen der ersten Förderungsperiode wurde besonders die lysosomale Degradation von HEV im endosomalen System als zentraler Ankerpunkt antiviraler Aktivität identifiziert. Nebst angeborener Immunantwort spielt hierbei die zelluläre Cholesterolhomöostase eine essenzielle Rolle. Basierend darauf, sollen zugrundeliegende Regulationen besser charakterisiert werden, um den Weg für Drug-Repurposing Verfahren zu bereiten und neue, assoziierte Zielstrukturen zu identifizieren. Eine Erweiterung der bisherigen Systeme soll via Tiermodell auch auf die adaptive Immunantwort abzielen, um ein breites, klinisches Bild zu erhalten.
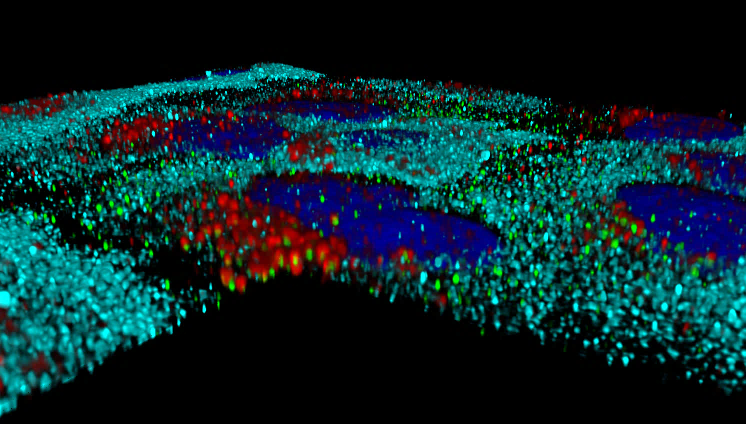
Dreidimensionale Rekonstruktion von HEV (grün) , GBP1 (cyan) und Lysosomen (rot) nach Behandlung mit Interferon .
Mittels CLSM erfasste, dreidimensionale Rekonstruktion von HEV (grün) in Lysosomen (cyan) nach Induktion einer Cholesterolakkumulation (magenta).
Wissenschaftliches Ziel:
Inhibition des endosomalen Lebenszyklus des HEV durch Modulation Cholesterol-abhängig regulierter Zielstrukturen mittels Applikation geeigneter Wirkstoffe („drug repurposing“).
DRUID-Kooperationspartner:
A2 Grünweller lab, A4 Heine/Reuter lab, B1 Diederich/Kolb lab, B6P Herker lab, C5 Glebe/Geyer lab, D1 Steinmetzer lab
Literatur D2: [1] Glitscher et al. (2018) Viruses 10(6):301; [2] Müller et al. (2020) Antiviral Res 174:104706; [3] Basic et al. (2019) Antiviral Res 172:104644; [4] Glitscher et al. (2021) J Virol doi: 10.1128/JVI.01564-20; [5] Glitscher et al. (2021) Cell Mol Gastroenterol Hepatol, doi:10.1016 /j.jcmgh.2021.02.002; [6] Himmelsbach et al. (2018) Emerg Microbes Infect 7(1):196. [7] Glitscher et al. (2021) Cell Microbiol. 16:e13379
D3Charakterisierung apoptotischer Parasiten zur Identifizierung neuer Zielmoleküle für die Behandlung von Leishmaniose
Ger van Zandbergen
Prof. Dr. Ger van Zandbergen
Abteilung Immunologie
Paul-Ehrlich-Institut
Paul-Ehrlich-Str. 51-59
63225 Langen
Tel.: +49 (0)6103-77 2005
E-Mail: Ger.Zandbergen(at)pei(dot)de
Projektbeschreibung
Leishmaniose ist eine vernachlässigte Tropenkrankheit, die durch den Parasiten Leishmania spp. verursacht wird und in fast 100 Ländern endemisch ist. Wir konnten zeigen, dass apoptotische Leishmania major (L. major) Promastigoten sowohl für die Infektiosität als auch das Überleben von Parasiten in den Wirtszellen verantwortlich sind. Apoptotische Parasiten induzieren eine anti-inflammatorische Antwort in humanen Makrophagen, die zu keiner effektiven T-Zellantwort gegen Leishmanien führt. Auch wenn L. major Parasiten alle typischen Apoptose-Merkmale zeigen, sind jedoch typische eukaryotische Apoptose-regulierende Proteine in Leishmanien nicht vorhanden.
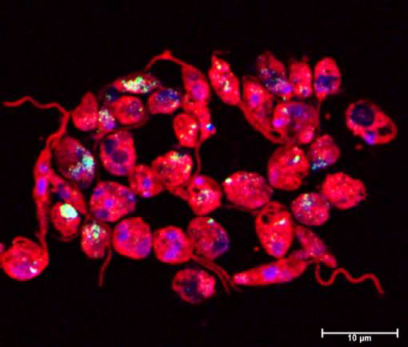
Färbung fragmentierter DNA (TUNEL staining) in L. major Cas9/T7, in denen durch Miltefosin Apoptose induziert wurde. (TUNEL: grün; DAPI: blau; anti-Lm-Serum: rot. ©Ger van Zandbergen)
Wissenschaftliches Ziel:
Für einen neuartigen Impfansatz und zur Tötung der Parasiten möchten wir zur Apoptose-regulierende Proteine in Leishmanien als mögliche neue Medikamenten-Targets identifizieren sowie attenuierte Leishmanien-Stämme ohne anti-inflammatorische Eigenschaften.
DRUID-Kooperationspartner:
B1 Kolb, B3 Rahlfs/Kolb, E3 Rahlfs/Przyborski
Literatur D3: 1. Arens et al. (2018) Front Immunol 31(9):1772. 2. Crauwels et al. (2019) Front Immunol 22(10):2697. Further publications within DRUID: 3. Turoňová et al. (2020) Science 370(6513):203-208.
D4Inhibition der Glutaminolyse und Glycolyse zur Hemmung der C. parvum-Infektion sowie One Health-Studie zur Kryptosporidiose in Kamerun
Carlos Hermosilla, Anja Taubert, Sybille Mazurek
Prof. Dr. Sybille Mazurek
Institut für Veterinär-Physiologie und -Biochemie
Fachbereich Veterinärmedizin
Justus-Liebig-Universität Gießen
Frankfurter Str. 100
35392 Gießen
Tel.: + 49 (0)641-99 38182
E-Mail Sybille.Mazurek(at)vetmed.uni-giessen(dot)de
Prof. Dr. Carlos Hermosilla
Institut für Parasitologie/Institut für Veterinär-Physiologie und -Biochemie
Justus-Liebig-Universität Gießen
Schubertstr. 81/Frankfurter Str. 100
Tel.: +49 (o)641-99 38461/99 38182
E-Mail: Carlos.R.Hermosilla(at)vetmed.uni-giessen(dot)de
Prof. Dr. Anja Taubert
BFS, Institut für Parasitologie
Justus-Liebig-Universität Gießen
Schubertstraße 81
35392 Gießen
Tel.: +49 (0)641-99 38460
Fax: +49 (0)641-99 38469
E-Mail: Anja.Taubert(at)vetmed.uni-giessen(dot)de
Projektbeschreibung
Kryptosporidien sind parasitäre Durchfallerreger beim Menschen, die insbesondere in Entwicklungsländern bei Kleinkindern und HIV-Infizierten zu hohen Morbiditäten und Todesfällen führen. Dabei sind die genauen infektionsbedingten Zusammenhänge in diversen Entwicklungsländern, wie z. B. Kamerun, nicht vollständig untersucht. Zur Therapie der Risikogruppen sind derzeit keine effektiven Medikamente erhältlich. Kryptosporidien sind obligat intrazelluläre Protozoen und verfügen selbst nur über minimale eigene Stoffwechselkapazitäten, daher müssen sie den Stoffwechsel der Wirtszelle zu ihrem Vorteil modulieren, um sich intrazellulär zu vermehren. Über die Charakterisierung der metabolischen Signaturen C. parvum-infizierter Wirtszellen konnten wir Stoffwechselreaktionen und -wege der Wirtzelle identifizieren, die für die Parasitenvermehrung essenziell sind.

Cryptosporidium parvum- (gelb) infizierte Wirtszellen (Zellkerne: blau), tomografische Mikroskopie © Juan Vélez
Hemmung von Cryptosporidium parvum über Stoffwechselinhibitoren (Vélez et al. 2021c)
Wissenschaftliches Ziel:
Das Projekt zielt darauf ab, strategische Angriffspunkte im Stoffwechsel der Wirtszelle (v. a. Glykolyse, Glutaminolyse) über neue Inhibitoren oder Kombinationsbehandlungen zu blockieren und damit indirekt die Parasitenentwicklung zu hemmen. Daneben wird eine One-Health-Studie zur Kryptosporidiose in Kamerun unter Berücksichtigung diverser epidemiologisch wichtiger Parameter durchgeführt.
DRUID-Kooperationspartner:
E4 Spengler lab
Literatur D4: 1. *Vélez et al. (2021a) Pathogens 11(1):49. 2. * Vélez et al. (2021b) Biology 10(10):963 3. ** Vélez et al. (2021c) Biology 10(1):60
Projektbereich E:
E1Plattform Wirkstofftestung gegen Helminthen
Simone Häberlein, Christoph Grevelding
PD Dr. Simone Häberlein
Justus-Liebig-Universität Gießen
BFS, Institut für Parasitologie
Schubertstraße 81
35392 Gießen
Tel.: +49 (0)641-99 38476
Fax: +49 (0)641-99 38469
E-Mail: Simone.Haeberlein(at)vetmed.uni-giessen(dot)de
Prof. Dr. Christoph Grevelding
BFS, Institut für Parasitologie
Justus-Liebig-Universität Gießen
Schubertstraße 81
35392 Gießen
Tel.: +49 (0)641-99 38466
Fax: +49 (0)641-99 38469
E-Mail: Christoph.Grevelding(at)vetmed.uni-giessen(dot)de
Projektbeschreibung
Targetstrukturen anti-parasitärer Substanzen sind oft konserviert, was die Möglichkeit der Therapie unterschiedlicher Parasitosen mit demselben Wirkstoff eröffnet. Im Plattformprojekt E1 fokussieren wir uns auf verschiedene parasitäre Würmer (Helminthen), für die eine Entwicklung neuer Wirkstoffe aufgrund von Resistenzen bzw. suboptimaler Therapieerfolge hohe Dringlichkeit hat. Dazu führen wir in vitro-Screenings von Substanzen aus DRUID-Projekten u.a. gegen den Bilharzioseerreger Schistosoma mansoni und den Leberegel Fasciola hepatica durch. Das Plattformprojekt verfügt über zahlreiche nationale und internationale Kooperationspartner und -partnerinnen, auch in endemischen Ländern in Asien und Afrika, die uns Testmöglichkeiten gegen eine Vielzahl weiterer Helminthenspezies ermöglichen.
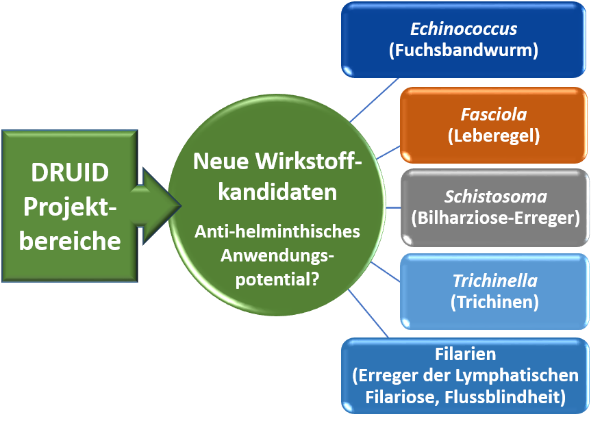
Strategie zur Identifizierung von Substanzen mit breitem Wirkspektrum gegenüber weltweit wichtige Helminthen-arten. ©Simone Häberlein
Für ausgewählte Substanzen werden zudem Wirkmechanismus-Analysen durchgeführt, wobei verschiedene in-vitro-Kultur-basierte und bildgebende Methoden zum Einsatz kommen.
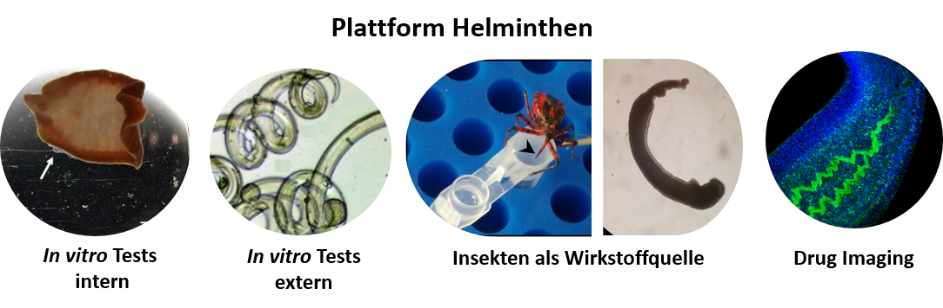
Testmöglichkeiten des Helminthen-Plattformprojekts für Wirkstoffkandidaten. ©Simone Häberlein, Miray Tonk-Rügen
Wissenschaftliches Ziel:
Ziel des Projekts ist die Identifizierung von Substanzen mit breitem anti-parasitären Wirkspektrum sowie die Aufklärung konservierter Wirkmechanismen.
DRUID-Kooperationspartner:
A2 Grünweller lab, A6 Douglas lab, A7 Przyborski lab, B4 Schlitzer lab, B5 Grevelding lab, B7 Falcone lab, E4 Spengler lab
Literatur E1: 1. Peter-Ventura et al. (2019) ChemMedChem 14(21):1856-1862. 2. Houhou et al. (2019) Sci Rep 9:15867. 3. Li et al. (2019) Parasitol Res 118(3):881-890. 4. Morawietz et al. (2020) Front Vet Sci 7:611270. 5. Kellershohn et al. (2019) PLOS Negl Trop Dis 13:e0007240. 6. Tonk et al. (2020) Antibiotics 9:664. 7. Mokosch et al. Anal Bioanal Chem 413(10): 2755-2766. 8. Mughal et al. (2021a) Int J Parasitol 51(7):571-585. 9. Mughal et al. (2021b) Int J Parasitol S0020-7519(21)00312-X. 10. Gallinger et al. (2022) Pharmaceuticals 15(2): 119. 11. Morawietz et al. (2022) Parasitol Res (online ahead of print) doi: 10.1007/s00436-021-07388-1
E3Proteinproduktion, HTS-Assays, Kristallisation, Interaktionsanalysen
Jude Przyborski, Stefan Rahlfs
Prof. Dr. Jude Przyborski
Interdisziplinäres Forschungszentrum (iFZ)
Justus-Liebig-Universität Gießen
Heinrich-Buff-Ring 26-32
35392 Gießen
Tel.: +49 (0)641-99 39114
E-Mail: jude.przyborski(at)ernaehrung.uni-giessen(dot)de
Dr. Stefan Rahlfs
Biochemie und Molekularbiologie
Justus-Liebig-Universität Gießen
Heinrich-Buff-Ring 26-32
35390 Gießen
Tel.: +49 (0)641-99 39117
E-Mail: stefan.rahlfs(at)ernaehrung.uni-giessen(dot)de
Projektbeschreibung
Rahmen der Plattform [E3] sollen Methoden zur Proteinproduktion, zur Entwicklung von high-throughput Assays, zur Kristallisation und Strukturanalyse sowie zu Interaktionsanalysen für das DRUID-Konsortium bereitgestellt werden. Dabei kann eine Vielzahl an Optimierungsmöglichkeiten bei der Klonierung/Expression sowie bei der Reinigung nativer Proteine (Affinitäts-chromatographie, Gelfiltration, Ionenaustauscher) angeboten werden. Für erfolgreich rekombinant produzierte Proteine kann über die Plattform [E3] Unterstützung bei der Entwicklung von Assaysystemen bis hin zu high throughput-fähigen Formaten gegeben werden. Des Weiteren soll über das Projekt [E3] Zugang zu der Kristallisationsplattform am iFZ ermöglicht werden. Für Kristallisationscreens im nl-Massstab stehen ein Pipettier- sowie ein Kristallisationsroboter sowie Starterkits und diverse Additivscreens zur Optimierung zur Verfügung, z. Z. wird das System durch Finanzierung von DRUID erneuert. Frau Dr. Fritz-Wolf wird als Kristallografin und Ansprechpartnerin fungieren und kann die ersten Testungen der Kristalle vornehmen. Hierzu stehen die Röntgenquellen des Max-Planck-Instituts für Med. Forschung (Heidelberg) bzw. der AG Klebe/Heine [A5] (Marburg) zur Verfügung.
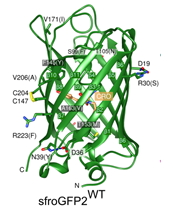
Struktur von sfroGFP (Heimsch et al. 2022)
DRUID-Kooperationspartner:
Im Bereich Protein-Expression: A1, A3, A6NWG, A7, B2, B3, B4, C3
Im Bereich Kristallisation: B2, B3, C3
Im Bereich Assay-Entwicklung: A7, B3
Literatur E3: 1. Heimsch et al. (2022) Antioxid & Redox Signal doi: 10.1089/ars.2021.0234 2. Harnischfeger et al. (2020) Electronic Journal of Biotechnology 3. Fritz-Wolf et al. (2011) Nat Commun 2:383 4. Koncarevic et al. (2009) Proc Natl Acad Sci USA 106:13323-8
E4Hochauflösende, bildgebende Massenspektrometrie
Bernhard Spengler
Prof. Dr. Bernhard Spengler
Institut für Anorganische und Analytische Chemie
Justus-Liebig-Universität Gießen
Heinrich-Buff-Ring 17
35392 Gießen
Tel.: +49 (0)641-99 34801
E-Mail: bernhard.spengler(at)ac.jlug(dot)de
Projektbeschreibung
Die hochauflösende, bildgebende Massenspektrometrie bietet vielfältige Einsatz–möglichkeiten, bei der Untersuchung von Pathogenen, der Pathogen-Wirts-Wechselwirkung sowie der Charakterisierung von Wirkstoffen. Die zu untersuchende Probe wird mithilfe eines Lasers abgerastert, die Schrittweite beträgt wenige Mikrometer. Hierbei wird Probenmaterial abgetragen und ionisiert, dies erlaubt die parallele Detektion hunderter chemischer Verbindungen sowie die bildliche Darstellung der Verteilung dieser Verbindungen in der Probe. Die Identifizierung der Moleküle erfolgt durch hochpräzise Messung der Ionenmasse in Verbindung mit Fragmentierungsexperimenten. Durch kontinuierliche Verbesserung der Ionisierungsmethode können immer kleinere Strukturen dargestellt und niedrig konzentrierte Analyten durch beispielweise in-situ Derivatisierung detektiert werden.
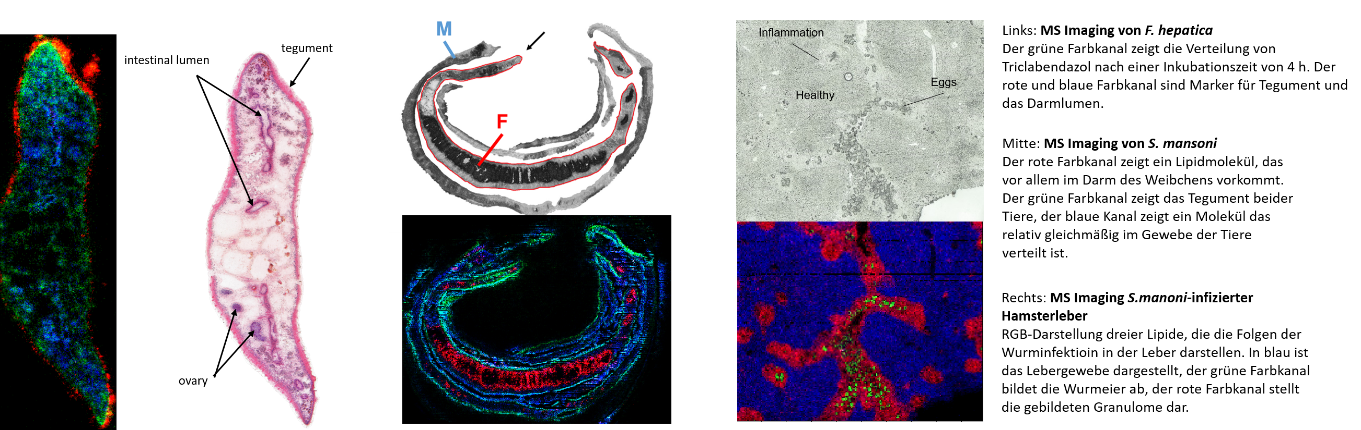
Wissenschaftliches Ziel:
Das Ziel des Projekts ist die Verbesserung und Anwendung der vorhandenen Methoden auf Fragestellungen innerhalb des DRUID Zentrums. Wichtige Forschungsfelder sind insbesondere die Verteilung von Wirkstoffen, lipidomische und metabolomische Charaktierisierung von Pathogenen und die Pathogen-Wirts-Interaktion.
DRUID-Kooperationspartner:
A2 Grünweller lab, B4 Grevelding lab, B5 Schlitzer lab, D4 Taubert lab, E1 Häberlein lab, W3 Herker lab
Literatur E4: [1] Kadesch et al. (2020) PLoS Negl. Trop. Dis. 14(5): e0008145; [2] Morawietz et al. (2020), Front. Vet. Sci. 7, 611270; [3] Mokosch et al. (2021) Anal Bioanal Chem 413, 2755–2766; [4] Müller et al. (2021) J. Am. Soc. Mass Spectrom.,3,32(2),465-472; [5] Spengler et al. (2015) Anal Chem 87 (1) 64-82.
E5Prozessentwicklung/-kontrolle (PAT, cGMP), Plattformen für die Produktion von Proteinen und Virusähnlichen Partikeln
Peter Czermak, Denise Salzig
Prof. Dr. Ing. Peter Czermak
Institut für Bioprozesstechnik
Technische Hochschule Mittelhessen
Wiesenstraße 14
35390 Gießen
Tel.: +49 (0)641-309 2551
E-Mail: peter.czermak(at)lse.thm(dot)de
Prof. Dr. Ing. Denise Salzig
Institut für Bioverfahrenstechnik
und Pharmazeutische Technologie
Technische Hochschule Mittelhessen
Gutfleischstr. 3-5
Tel.: +49 (0)641-309 2630
Fax: +49 (0)641-309 2553
E-Mail: denise.salzig(at)lse.thm(dot)de
Projektbeschreibung
Um Forschungsergebnisse aus dem DRUID Konsortium in die Klinik oder die Industrie zu translatieren, sind robuste Produktionsprozesse, die mehr als eine bloße Maßstabsvergrößerung darstellen, unabdingbar. Dabei müssen diese Prozesse den Richtlinien der guten Herstellungspraxis (cGMP) und der Prozessanalytischen Technologie (PAT) folgen. Folgende Arbeiten sind geplant: i) Prozessintensivierung und Ausbau der BEVS-Produktionsplattform sowie Umsetzung einer kontinuierlichen Prozessführung, ii) Einbindung von online PAT-Technologie, z.B. Impedanzspektroskopie zur automatischen Bestimmung des optimalen Erntezeitpunkts und der automatisierten Ernte, iii) Expression (BEVS) und Aufreinigung von S. mansoni-Kinasen sowie Untersuchung der Kinasen und putativer Inhibitoren, und iv) Untersuchung neuartiger Transfektionsreagenzien für die transiente Proteinproduktion im Bioreaktormaßstab.
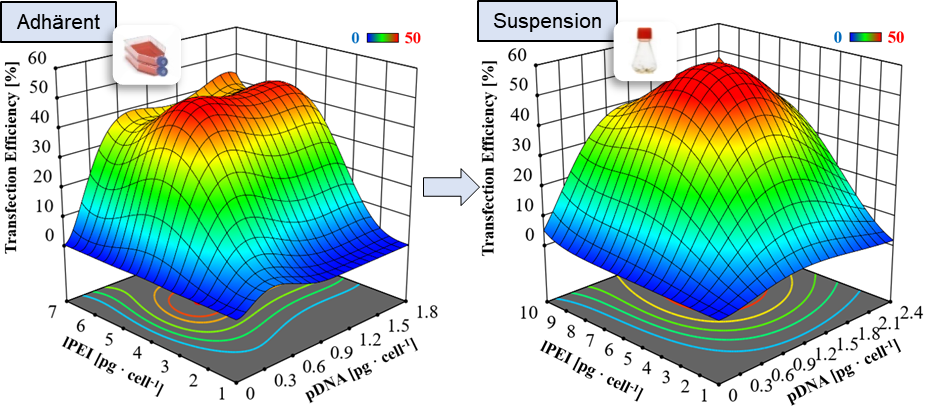
Optimierung des transienten Transfektionsprozesses mittels statistischer Versuchsplanung. ©IBPT
Produktionskonzept mit integrierter PAT-Technologie. ©IBPT
Wissenschaftliches Ziel:
Die Arbeiten zur Produktionsprozessentwicklung und Prozessanalytik werden weiter an die Fragestellungen des Zentrums angepasst, erweitert, und dem gesamten Konsortium zur Nutzung angeboten. Insbesondere ist geplant, die aufgebaute BEVS (Baculovirus-Expression-System)-Plattform weiter zu intensivieren und zu automatisieren. Darüber hinaus wird auch die zweite Produktionsplattform – die transiente Produktion mit HEK-293T-Zellen – weiterentwickelt.
DRUID-Kooperationspartner:
B4 AG Schlitzer, B5 AG Grevelding, B7 P AG Falcone, C6 NWG Häberlein, E1 Plattform Grevelding/Häberlein
Literatur E5: Biotechnol, DOI: 10.1016/j.ejbt.2021.08.002, 3. Lothert et al. (2020) Methods Mol Biol 2183:217-248, 4. Dekevic et al. (2022) J Biotechn 346 23-34, 5. Schwarz et al. (2021) Elec J Biotechnol, DOI: 10.1016/j.ejbt.2022.01.003; 6. Barekzai et al. (2020) New Adv Ferm Processes, DOI: 10.5772/intechopen.90029, 7. Eckhardt et al. (2021) Sep Sci Technol, 57 (6) 886-897
E6Pharmazeutische Entwicklung, Translationale Medizin, drug repurposing
Susanne Schiffmann
Dr. Susanne Schiffmann
Fraunhofer Institut für Translationale Medizin und Pharmakologie ITMP
Theodor-Stern-Kai 7
60596 Frankfurt am Main
Tel.: +49 (0)69-8700 25060
E-Mail: susanne.schiffmann(at)itmp.fraunhofer(dot)de
Projektbeschreibung
Die SARS-CoV2-Pandemie verdeutlicht, dass die Entwicklung von Arzneimitteln, die Infektionen therapieren können, dringend notwendig ist. Um neue Behandlungsoptionen zu identifizieren, werden vielversprechende Substanzen aus anderen Arbeitsgruppen präklinisch charakterisiert. Dies umfasst die Erstellung eines Sicherheitsprofils sowie die Bioverfügbarkeitstestung der Wirkstoffkandidaten. Da eine unterstützende Therapie mit immunmodulierenden Arzneimitteln einen bewährten Therapieansatz darstellt, wird eine Interaktion der Wirkstoffkandidaten mit dem Immunsystem geprüft.
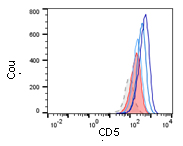
Veränderung der Oberflächenmarker-expression auf behandelten M2-Makrophagen ©Marina Henke
Veränderung der Zytokinfreisetzung in behandelten M2-Makrophagen ©Leonard Blum
Wissenschaftliches Ziel:
Im Rahmen des Projektes soll für small molecules/Peptide, die potenziell eine antivirale, antiparasitäre oder antibakterielle Wirkung haben, ein Sicherheits- und/oder immunmodulatorisches Profil erstellt werden. Des Weiteren werden in diesem Projekt humane Zielproteine (z. B. TMPRSS2) untersucht, die neue Targets zur Bekämpfung von Pathogenen darstellen.
DRUID-Kooperationspartner:
A1 Becker lab, A2 Grünweller lab, A3 Weber lab, B1 Diederich/Kolb lab, B3 Rahlfs/Kolb/van Zandbergen, B5 Schlitzer lab, D1 Friebertshäuser lab
Literatur E6: 1. Blum et al. (2021) J Mol Med 99(2):261-72; 2. Blum et al. (2020) Sci Rep 10(1):7534; 3. Blum et al. (2020) J Cell Mol Med 24(12):6988-99.
E7 PVernachlässigte Infektionskrankheiten mit Schwerpunkt bildgebender Verfahren
Jacomina Krijnse Locker
Prof. Dr. Jacomina Krijnse Locker
Elektronenmikroskopie von Pathogenen
Paul-Ehrlich-Institut
Paul-Ehrlich-Straße 51-59
63225 Langen
Tel.: +49 (0)6103-77 2011
E-Mail: Jacomina.KrijnseLocker(at)pei(dot)de
Projektbeschreibung
Wir setzen bildgebende Verfahren ein, um zu verstehen wie Krankheitserreger mit der Wirtszelle interagieren und Krankheiten verursachen. Unser Fokus liegt auf der Elektronenmikroskopie (EM) und korrelativer Licht- und Elektronenmikroskopie (CLEM). Um robuste, anpassbare Protokolle zu generieren, verwenden wir das große DNA-Virus Vaccinia (VACV) als Modell. Die SARS-CoV2-Pandemie veranschaulicht, wie Viren weltweit sozioökonomische Auswirkungen mit dramatischen Folgen für die vernachlässigten Tropenkrankheiten (NTDs) haben. Es besteht die dringende Notwendigkeit, die SARS-CoV2-Pandemie einzudämmen und die Ressourcen auf NTDs zu konzentrieren. In einer Instituts-übergreifenden Kooperation haben wir gezeigt, dass das Spike- Protein (S) von SARS-CO-2, das essenziell für die Infektion ist, eine unerwartete Flexibilität aufweist. Dies könnte die Rezeptorbindung und den Zelleintritt des Virus erleichtern, was bis jetzt nicht bewiesen ist.
Die Kryoelektronentomographie von SARS-CoV-2 und die molekulare dynamische Simulation des Spike Proteins zeigen eine unerwartete Flexibilität des Stiels.
Nach der Knospung (Entstehung der Virushülle) an der intrazellulären Membranen verlassen Coronaviren die Zelle auf eine bisher wenig erforschte Weise. Vesikuläres Trafficking sowie eine regulierte lysosomale Exozytose werden dabei als Mechanismen diskutiert.
Wissenschaftliches Ziel:
Ziel des Projektes ist es, mit Hilfe von Antikörpern, Medikamenten und Genetik in die Flexibilität von S einzugreifen, Auswirkungen auf die Infektion zu testen und damit die Funktion zu verstehen. Die Virusfreisetzung aus infizierten Zellen soll mit einem CLEM-Ansatz untersucht werden, der Live-Cell-Imaging mit EM kombiniert. Die aus unserem VACV- und SARS CoV2-Wirtssystem gewonnene Expertise kann dann zur Beantwortung von Fragen im Kontext anderer Virus-Wirtssystemen der Teams des DRUID-Konsortiums eingesetzt werden.
DRUID-Kooperationspartner:
A3 Weber lab, B2 Ziebuhr lab, B6 P Herker lab, C1 Hildt lab, D3 van Zandbergen lab, E5 Czermak lab
Literatur E7 P: Turoňová, B., et al. (2020). Science (80-. ). 370, 203–208. Blanco-Rodriguez, G., et al.(2020). J. Virol. 94 e00135-20. Quemin, E.R., et al. (2018). J. Mol. Biol.430, 1714-1724. Chlanda, P., and Krijnse Locker, J. (2017). Biochem. J. 474, 1041–1053. Sartori-Rupp, A., et al. (2019). Nat. Commun. 10, 342.


































