A2: Entwicklung von eIF4A-Inhibitoren als Kandidaten für eine klinische Anwendung sowie Charakterisierung von eIF4A-Varianten in Pathogenen
Prof. Dr. Arnold Grünweller
Philipps-Universität Marburg
Bau C
Marbacher Weg 6
35032 Marburg
Tel.: +49 (0)6421-2825849
Fax: +49 (0)6421-28 25854
E-Mail: arnold.gruenweller(at)staff.uni-marburg(dot)de
Projektbeschreibung
Die zelleigene RNA-Helikase eIF4A ist ein exzellentes Target für die Entwicklung antiviraler Breitbandmedikamente. Dieses Enzym wird für den Start der viralen Proteinsynthese von vielen Viren, insbesondere Coronaviren, benötigt und kann effizient und spezifisch durch die Stoffgruppe der Rocaglate gehemmt werden. In Projekt A2 möchten wir entsprechende Wirkstoffe für eine klinische Anwendung weiterentwickeln, indem wir unter anderem Rocaglate für eine lokale Applikation in den respiratorischen Trakt vernebeln und ein detailliertes Nebenwirkungsprofil erstellen. Zudem sollen neue eIF4A-Inhibitoren identifiziert und charakterisiert werden. Durch systematische Mutagenese bekannter Coronavirus-Sequenzen soll außerdem eine Vorhersage der Rocaglat-Sensitivität bei neu auftretenden Coronaviren ermöglicht werden. Schließlich soll die therapeutische Relevanz von Rocaglaten in verschiedenen DRUID-relevanten Pathogenen, die Varianten von eIF4A exprimieren, untersucht werden.
![]()
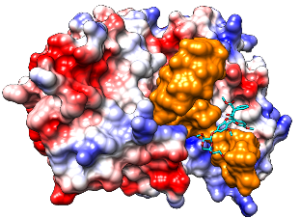

Festklemmen der RNA (RNA clamping) an der Oberfläche von eIF4A durch das Rocaglat Silvestrol sowie Nachweis der Hemmung der (corona)viralen Proteinsynthese durch das Rocaglat CR-31-B (-).
Wissenschaftliches Ziel:
Rocaglate sollen für eine klinische Anwendung weiterentwickelt und eine Vorhersage der Rocaglat-Sensitivität bei neu auftretenden Coronaviren und anderen DRUID-relevanten Pathogenen ermöglicht werden.
DRUID-Kooperationspartner:
B2 Ziebuhr lab, B4 Grevelding lab, B5 Schlitzer lab, C6 Häberlein lab, D1 Friebertshäuser/Steinmetzer lab, D3 van Zandbergen lab, D4 Hermosilla/Mazurek/Taubert lab, E4 Spengler lab, E6 Schiffmann lab
Literatur A2: 1.*Biedenkopf et al., (2017), Antiviral Res. 137: 76-81; 2. *Müller et al., (2018), Antiviral Res. 150:123-129; 3. *Elgner et al., (2018), Viruses. 10(4): 149; 4. *Glitscher et al., (2018), Viruses. 10(6): 301; 5. *Henß et al., (2018), Viruses. 10(11): 592; 6. *Müller et al., (2020), Antiviral Res. 175:104706; *7. *Müller et al., (2021), Antiviral Res. 186: 105012; 8. *Blum et al., (2020), J Cell Mol Med. 24(12): 6988-6999; [*eigene Publikationen].

