Projektübersicht
Prof. Dr. Peter Kolb
Institut für Pharmazeutische Chemie
Philipps-Universität Marburg
Marbacher Weg 8
35032 Marburg
Tel.: +49 (0)6421-28 25908
Fax: +49 (0)6421-28 26652
E-Mail: peter.kolb(at)uni-marburg(dot)de

NAD(P)H-abhängige Stoffwechselwege als Ziele für neue Antiinfektiva
Projektbeschreibung
Das zelluläre Redoxgleichgewicht spielt für pathogene Mikroorganismen eine essentielle Rolle. Zentral daran beteiligt sind Enzyme des NAD(P)H-abhängigen Glutathion- und Thioredoxinsystems1,2, sowie die Glucose-6-Phosphatdehydrogenase (G6PD) und 6-Phosphogluconatdehydrogenase (6PGD), die maßgeblich zur Bereitstellung von NADPH und Ribose-5-Phosphat über den Pentosephosphatweg beitragen3. Die bifunktionale G6PD der Malariaparasiten Plasmodium falciparum und P. vivax (GluPho) unterscheidet sich funktionell und strukturell von den humanen Wirtsenzymen4 und ist essenziell für den Malariaparasiten5. In Kooperation mit dem Sanford-Burnham Institute/UCSD, La Jolla, konnten wir für die PfGluPho einen high-throughput kompatiblen Assay etablieren und ca. 400.000 Substanzen (u.a. NIH MLSMR Collection) screenen6. Anschließende Struktur-Wirkungs-Analysen und Lead-Optimierung resultierten in den im nanomolaren Bereich aktiven und hoch selektiven PfGluPho Inhibitor SBI-7507. Das Konzept konnte bereits auf Leishmania übertragen werden, die 3D-Kristallstruktur der Leishmania donovani G6PD und PGD gelöst werden und auf deren Basis ein erstes in silico Screening von Kleinmolekülen durchgeführt werden. Darüber hinaus wurde ein high-throughput kompatibler Assay für die rekombinanten G6PDs und 6PGDs aus Leishmania etabliert, um ein Inhibitor-Screening bei dem Novartis FAST lab in Cambridge (USA) durchzuführen.
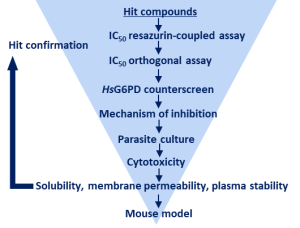
Workflow-Diagramm zur Inhibitor Identifizierung gegen G6PDs/PGDs
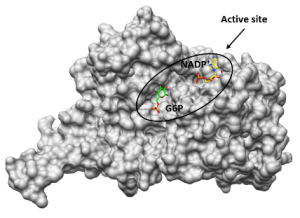
3D-Kristallstruktur LdG6PD ©Isabell Berneburg
Wissenschaftliches Ziel:
Ziel des Projektes ist die funktionelle und strukturelle Charakterisierung der Enzyme G6PD und 6PGD aus Leishmania und Plasmodium als Zielmoleküle für die Wirkstoffentwicklung, sowie die Identifizierung und Weiterentwicklung von Inhibitoren dieser Enzyme (in silico und HTS). Das Konzept soll auch auf weitere Erreger innerhalb des DRUID Konsortiums, wie z.B. Schistosoma, übertragen werden.
DRUID-Kooperationspartner:
B1 Diederich/Kolb; B4 Grevelding; D3 van Zandbergen; E1 Grevelding/Häberlein; E3 Rahlfs/Przyborski; E4 Spengler; E6 Schiffmann/Laux
Literatur B3: 1. Fritz-Wolf et al. (2011) Nature Comm. 2:383* 2. Koncarevic et al. (2009) PNAS 106: 13323-8* 3. Bozdech and Ginsburg (2005) Malaria J 3:23 4. Jortzik et al. (2011) Biochem J Energy 436:641-50* 5. Allen et al. (2015) FEBS J 282:3808-23*, 6. Preuss et al. (2012) J Med Chem 55:7262-72* 7. Berneburg et al. (2022) Antimicrob Agents Chemother (accepted)*
*eigene Publikationen
Dengue- und Zikavirus, Design von Inhibitoren der NS3/NS2B-Serinprotease
Projektbeschreibung
Infektionen mit dem Dengue-Virus haben in den letzten Jahren mit rund 50-100 Millionen Neuinfektionen pro Jahr einen neuen Höchststand erreicht. In der überwiegenden Zahl der Fälle verläuft die Infektion mit milden, grippeähnlichen Symptomen, jedoch entwickelt ein kleiner Prozentsatz der Betroffenen, häufig Kinder, ein hämorrhagisches Fieber, das bei schwerem Verlauf tödlich verläuft. Zwar steht inzwischen ein Impfstoff zur Verfügung, da dieser aber nur für einen sehr begrenzten Personenkreis zugelassen ist, ist die Entwicklung von Wirkstoffen, die die Vermehrung des Virus effizient unterdrücken, unabdingbar. Im Fokus unser Arbeiten steht die viruseigene Serinprotease NS3/NS2B, die das virale Vorläuferprotein in funktionelle Proteine spaltet und für Reifung des Virus unerlässlich ist.
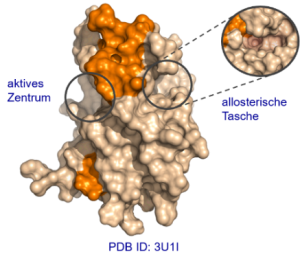
Abb. B1. Kristallstruktur der NS3/NS2B Protease DENV 3.1
Wissenschaftliches Ziel:
Ziel des Projektes ist die Weiterentwicklung allosterischer Inhibitoren der viruseigenen Serinprotease NS3/NS2B, nicht nur hinsichtlich ihrer Affinität, sondern auch in Bezug auf ihre pharmakokinetischen Eigenschaften und ihre Toxizität (hit-to-lead-Entwicklung) mittels eines kombinierten Ansatzes aus computergestütztem Design, Synthese, biologischen Tests und der Kristallstrukturanalyse. Darüber hinaus sollen basierend auf den gewonnenen Erkenntnisse Inhibitoren der verwandten Zika-Protease entwickelt werden.
DRUID-Kooperationspartner:
A1 Becker, A2 Grünweller, B3 Rahlfs/Kolb/van Zandbergen, XY Herker, E6 Schiffmann/Laux
Literatur B1: [1] Noble et al. (2012) J Virol 86(1):438-446; *[2] Chevillard et al. (2015), J Chem Inf Model 55(9):1824-1835; *[3] Chevillard et al. (2018) J Med Chem, 61(3):1118-1129.
