Projektübersicht
Prof. Dr. Ger van Zandbergen
Abteilung Immunologie
Paul-Ehrlich-Institut
Paul-Ehrlich-Str. 51-59
63225 Langen
Tel.: +49 (0)6103-77 2005
E-Mail: Ger.Zandbergen(at)pei(dot)de

Charakterisierung apoptotischer Parasiten zur Identifizierung neuer Zielmoleküle für die Behandlung von Leishmaniose
Projektbeschreibung
Leishmaniose ist eine vernachlässigte Tropenkrankheit, die durch den Parasiten Leishmania spp. verursacht wird und in fast 100 Ländern endemisch ist. Wir konnten zeigen, dass apoptotische Leishmania major (L. major) Promastigoten sowohl für die Infektiosität als auch das Überleben von Parasiten in den Wirtszellen verantwortlich sind. Apoptotische Parasiten induzieren eine anti-inflammatorische Antwort in humanen Makrophagen, die zu keiner effektiven T-Zellantwort gegen Leishmanien führt. Auch wenn L. major Parasiten alle typischen Apoptose-Merkmale zeigen, sind jedoch typische eukaryotische Apoptose-regulierende Proteine in Leishmanien nicht vorhanden.
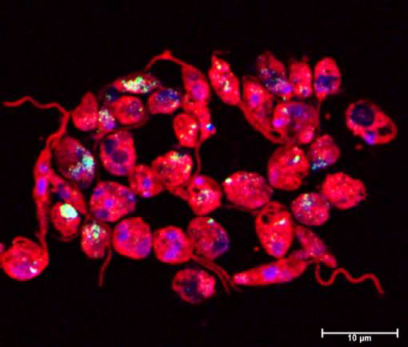
Färbung fragmentierter DNA (TUNEL staining) in L. major Cas9/T7, in denen durch Miltefosin Apoptose induziert wurde. (TUNEL: grün; DAPI: blau; anti-Lm-Serum: rot. ©Ger van Zandbergen)
Wissenschaftliches Ziel:
Für einen neuartigen Impfansatz und zur Tötung der Parasiten möchten wir zur Apoptose-regulierende Proteine in Leishmanien als mögliche neue Medikamenten-Targets identifizieren sowie attenuierte Leishmanien-Stämme ohne anti-inflammatorische Eigenschaften.
DRUID-Kooperationspartner:
B1 Kolb, B3 Rahlfs/Kolb, E3 Rahlfs/Przyborski
Literatur D3: 1. Arens et al. (2018) Front Immunol 31(9):1772. 2. Crauwels et al. (2019) Front Immunol 22(10):2697. Further publications within DRUID: 3. Turoňová et al. (2020) Science 370(6513):203-208.
NAD(P)H-abhängige Stoffwechselwege als Ziele für neue Antiinfektiva
Projektbeschreibung
Das zelluläre Redoxgleichgewicht spielt für pathogene Mikroorganismen eine essentielle Rolle. Zentral daran beteiligt sind Enzyme des NAD(P)H-abhängigen Glutathion- und Thioredoxinsystems1,2, sowie die Glucose-6-Phosphatdehydrogenase (G6PD) und 6-Phosphogluconatdehydrogenase (6PGD), die maßgeblich zur Bereitstellung von NADPH und Ribose-5-Phosphat über den Pentosephosphatweg beitragen3. Die bifunktionale G6PD der Malariaparasiten Plasmodium falciparum und P. vivax (GluPho) unterscheidet sich funktionell und strukturell von den humanen Wirtsenzymen4 und ist essenziell für den Malariaparasiten5. In Kooperation mit dem Sanford-Burnham Institute/UCSD, La Jolla, konnten wir für die PfGluPho einen high-throughput kompatiblen Assay etablieren und ca. 400.000 Substanzen (u.a. NIH MLSMR Collection) screenen6. Anschließende Struktur-Wirkungs-Analysen und Lead-Optimierung resultierten in den im nanomolaren Bereich aktiven und hoch selektiven PfGluPho Inhibitor SBI-7507. Das Konzept konnte bereits auf Leishmania übertragen werden, die 3D-Kristallstruktur der Leishmania donovani G6PD und PGD gelöst werden und auf deren Basis ein erstes in silico Screening von Kleinmolekülen durchgeführt werden. Darüber hinaus wurde ein high-throughput kompatibler Assay für die rekombinanten G6PDs und 6PGDs aus Leishmania etabliert, um ein Inhibitor-Screening bei dem Novartis FAST lab in Cambridge (USA) durchzuführen.
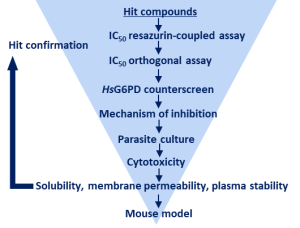
Workflow-Diagramm zur Inhibitor Identifizierung gegen G6PDs/PGDs
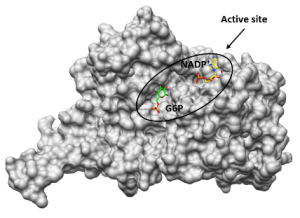
3D-Kristallstruktur LdG6PD ©Isabell Berneburg
Wissenschaftliches Ziel:
Ziel des Projektes ist die funktionelle und strukturelle Charakterisierung der Enzyme G6PD und 6PGD aus Leishmania und Plasmodium als Zielmoleküle für die Wirkstoffentwicklung, sowie die Identifizierung und Weiterentwicklung von Inhibitoren dieser Enzyme (in silico und HTS). Das Konzept soll auch auf weitere Erreger innerhalb des DRUID Konsortiums, wie z.B. Schistosoma, übertragen werden.
DRUID-Kooperationspartner:
B1 Diederich/Kolb; B4 Grevelding; D3 van Zandbergen; E1 Grevelding/Häberlein; E3 Rahlfs/Przyborski; E4 Spengler; E6 Schiffmann/Laux
Literatur B3: 1. Fritz-Wolf et al. (2011) Nature Comm. 2:383* 2. Koncarevic et al. (2009) PNAS 106: 13323-8* 3. Bozdech and Ginsburg (2005) Malaria J 3:23 4. Jortzik et al. (2011) Biochem J Energy 436:641-50* 5. Allen et al. (2015) FEBS J 282:3808-23*, 6. Preuss et al. (2012) J Med Chem 55:7262-72* 7. Berneburg et al. (2022) Antimicrob Agents Chemother (accepted)*
*eigene Publikationen
