Projektübersicht
Prof. Dr. Eberhard Hildt
Bundesinstitut für Impfstoffe
und biomedizinische Arzneimittel
Paul-Ehrlich-Institut
Paul-Ehrlich-Straße 51-59
63225 Langen
Tel.: +49 (0)6103-77 2140
Fax: +49 (0)6103-77 1234
E-Mail: Eberhard.Hildt(at)pei(dot)de

HEV, Identifizierung zellulärer Targets für antivirale Strategien – Hemmung der Virusfreisetzung durch Modulation des Cholesterolspiegels
Projektbeschreibung
Das Hepatitis E Virus führt jährlich zu über 20 Mio. Infektionen weltweit und betrifft Industrienationen, besonders allerdings Entwicklungsländer. Limitierte Behandlungs-optionen sind häufig mit schweren Nebenwirkungen verbunden. Da das unbehüllte Virus für seine Freisetzung endosomale Prozesse nutzt, bietet dies einen vielversprechenden Ansatzpunkt für neue Wirkstoffe. Im Rahmen der ersten Förderungsperiode wurde besonders die lysosomale Degradation von HEV im endosomalen System als zentraler Ankerpunkt antiviraler Aktivität identifiziert. Nebst angeborener Immunantwort spielt hierbei die zelluläre Cholesterolhomöostase eine essenzielle Rolle. Basierend darauf, sollen zugrundeliegende Regulationen besser charakterisiert werden, um den Weg für Drug-Repurposing Verfahren zu bereiten und neue, assoziierte Zielstrukturen zu identifizieren. Eine Erweiterung der bisherigen Systeme soll via Tiermodell auch auf die adaptive Immunantwort abzielen, um ein breites, klinisches Bild zu erhalten.
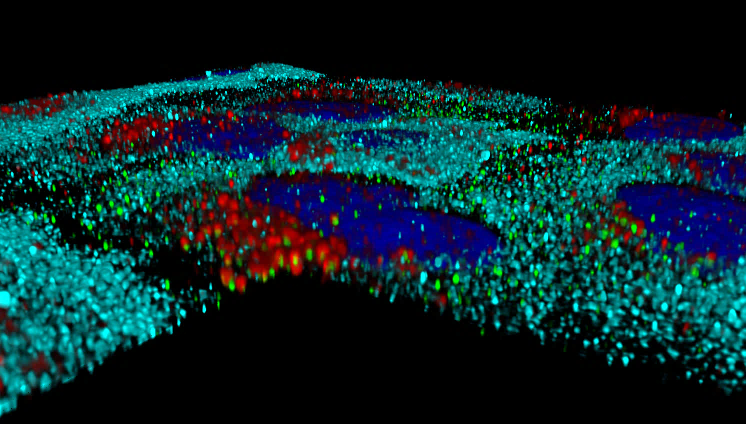
Dreidimensionale Rekonstruktion von HEV (grün) , GBP1 (cyan) und Lysosomen (rot) nach Behandlung mit Interferon .
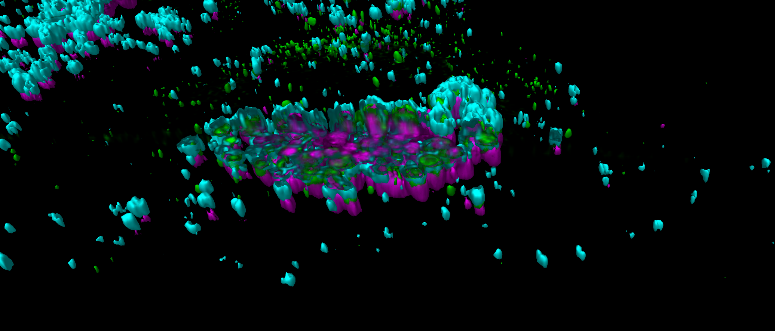
Mittels CLSM erfasste, dreidimensionale Rekonstruktion von HEV (grün) in Lysosomen (cyan) nach Induktion einer Cholesterolakkumulation (magenta).
Wissenschaftliches Ziel:
Inhibition des endosomalen Lebenszyklus des HEV durch Modulation Cholesterol-abhängig regulierter Zielstrukturen mittels Applikation geeigneter Wirkstoffe („drug repurposing“).
DRUID-Kooperationspartner:
A2 Grünweller lab, A4 Heine/Reuter lab, B1 Diederich/Kolb lab, B6P Herker lab, C5 Glebe/Geyer lab, D1 Steinmetzer lab
Literatur D2: [1] Glitscher et al. (2018) Viruses 10(6):301; [2] Müller et al. (2020) Antiviral Res 174:104706; [3] Basic et al. (2019) Antiviral Res 172:104644; [4] Glitscher et al. (2021) J Virol doi: 10.1128/JVI.01564-20; [5] Glitscher et al. (2021) Cell Mol Gastroenterol Hepatol, doi:10.1016 /j.jcmgh.2021.02.002; [6] Himmelsbach et al. (2018) Emerg Microbes Infect 7(1):196. [7] Glitscher et al. (2021) Cell Microbiol. 16:e13379
Targets für antivirale Strategien gegen das Zika Virus
Projektbeschreibung
Zika-Viren (ZIKV) sind Arboviren aus der Familie der Flaviviren. Im Zuge der ZIKV Epidemie 2015/2016 hat die WHO am 01.02.2016 den Ausbruch in Südamerika zum Public Health Emergency of International Concern (PHEIC) erklärt, da es einen Zusammenhang zwischen der Zika-Virus-Infektion Schwangerer und dem Auftreten von Mikrozephalien bei Neugeborenen gibt. Derzeit steht weder eine Impfung noch eine spez. Therapie zur Verfügung. Durch Hemmung der Virusreplikation bereits in der frühen Phase der Infektion ggf. mittels einer temporären präventiven Strategie könnte die Viruslast deutlich reduziert und so die Verbreitung des Virus und so auch das Risiko einer intrauterinen Infektion vermindert werden. Im Rahmen der ersten Förderperiode konnten Zielstrukturen für antivirale Ansätze identifiziert werden.
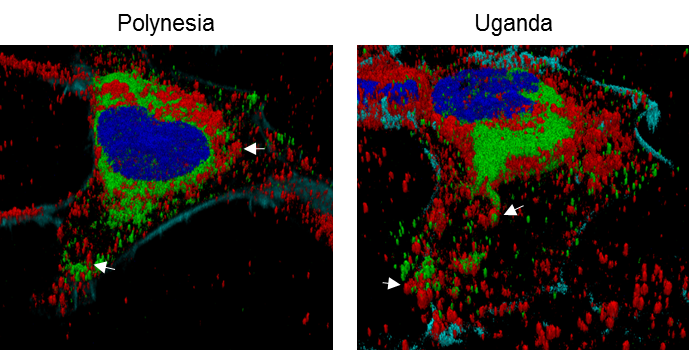
Intrazelluäre Verteilung von Tetherin (rot) und dem ZIKV Hüllprotein E (grün) in Zellen infiziert mit zwei verschiedenen ZIKV Isolaten (Uganda bzw. Polynesia)
Wissenschaftliches Ziel:
Basierend auf bereits identifizierten Zielstrukturen und weiterer Zielstrukturen sollen antivirale Strategien entwickelt, der Mechanismus charakterisiert und deren Wirkung auch auf weitere Flaviviren untersucht werden.
DRUID-Kooperationspartner:
A2 AG Grünweller, B1 AG Diderich/Kolb, B6P AG Herker, C2 AG Kempf, C5 AG Glebe/Geyer, D1 AG Steinmetzer, E6 AG Schiffmann/Laux
Literatur C1: 1. Herrlein et al. (2021) J Virol. doi: 10.1128/jvi.02117-2 2. Sabino et al (2021)., J Virol. doi: 10.1128 3. *Maddaluno et al., 2020 EMBO Mol Med. doi: 10.15252/emmm.201911793 4. Basic et al. 2019 Antiviral Res. doi: 10.1016/j.antiviral.2019.104644. 5. Akhras et al. (2019) Viruses doi: 10.3390/v11080748. 6. Sabino et al., (2019) doi: 10.3390/v11060524 7. Elgner et al (2018) Viruses doi: 10.3390/v10040149
