Projektübersicht
Prof. Dr. Jude Przyborski
Interdisziplinäres Forschungszentrum (iFZ)
Justus-Liebig-Universität Gießen
Heinrich-Buff-Ring 26-32
35392 Gießen
Tel.: +49 (0)641-99 39114
E-Mail: jude.przyborski(at)ernaehrung.uni-giessen(dot)de

Proteinproduktion, HTS-Assays, Kristallisation, Interaktionsanalysen
Projektbeschreibung
Rahmen der Plattform [E3] sollen Methoden zur Proteinproduktion, zur Entwicklung von high-throughput Assays, zur Kristallisation und Strukturanalyse sowie zu Interaktionsanalysen für das DRUID-Konsortium bereitgestellt werden. Dabei kann eine Vielzahl an Optimierungsmöglichkeiten bei der Klonierung/Expression sowie bei der Reinigung nativer Proteine (Affinitäts-chromatographie, Gelfiltration, Ionenaustauscher) angeboten werden. Für erfolgreich rekombinant produzierte Proteine kann über die Plattform [E3] Unterstützung bei der Entwicklung von Assaysystemen bis hin zu high throughput-fähigen Formaten gegeben werden. Des Weiteren soll über das Projekt [E3] Zugang zu der Kristallisationsplattform am iFZ ermöglicht werden. Für Kristallisationscreens im nl-Massstab stehen ein Pipettier- sowie ein Kristallisationsroboter sowie Starterkits und diverse Additivscreens zur Optimierung zur Verfügung, z. Z. wird das System durch Finanzierung von DRUID erneuert. Frau Dr. Fritz-Wolf wird als Kristallografin und Ansprechpartnerin fungieren und kann die ersten Testungen der Kristalle vornehmen. Hierzu stehen die Röntgenquellen des Max-Planck-Instituts für Med. Forschung (Heidelberg) bzw. der AG Klebe/Heine [A5] (Marburg) zur Verfügung.
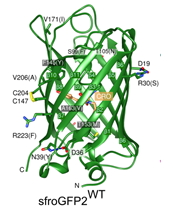
Struktur von sfroGFP (Heimsch et al. 2022)
DRUID-Kooperationspartner:
Im Bereich Protein-Expression: A1, A3, A6NWG, A7, B2, B3, B4, C3
Im Bereich Kristallisation: B2, B3, C3
Im Bereich Assay-Entwicklung: A7, B3
Literatur E3: 1. Heimsch et al. (2022) Antioxid & Redox Signal doi: 10.1089/ars.2021.0234 2. Harnischfeger et al. (2020) Electronic Journal of Biotechnology 3. Fritz-Wolf et al. (2011) Nat Commun 2:383 4. Koncarevic et al. (2009) Proc Natl Acad Sci USA 106:13323-8
Plasmodium falciparum Chaperone, Co-Chaperone und deren Protein-Protein-Interaktionen als Ziele für neue Interventionsstrategien
Projektbeschreibung
Übersicht der Assay-Entwicklung
Malariaparasiten dringen in reife menschliche rote Blutkörperchen (RBC) ein und leben darin. Um sein Überleben zu sichern, baut der Parasit die von ihm gewählte Wirtszelle zu seinem eigenen Vorteil um. Infizierte rote Blutkörperchen werden klebrig und haften an der Innenwand kleiner Blutgefäße, außerdem überziehen sie sich mit Proteinen, die es ihnen ermöglichen, für das Immunsystem unsichtbar zu werden. Dies führt leider zur Krankheit des Patienten und schließlich zum Tod. Wir haben kürzlich eine Reihe wichtiger molekularer Akteure identifiziert, die für diesen Umbauprozess wesentlich sind, darunter Mitglieder der so genannten HSP70- und HSP40-Familien. Das Ziel dieses Projekts ist es, die Funktion von HSP40 und HSP70 zu blockieren. Wenn uns dies gelingt, werden die Parasiten wahrscheinlich aus dem Blutkreislauf entfernt, wodurch die Schwere der Krankheit gelindert wird. Zu diesem Zweck werden wir eine Reihe von Assays zur Messung der Aktivität von HSP40/HSP70 entwickeln und auf dieser Grundlage nach Verbindungen suchen, die diese Wechselwirkung verringern. Vielversprechende Verbindungen werden dann direkt an Parasiten auf ihre Fähigkeit getestet, die Veränderung der Wirtszellen zu verringern.
Literatur A7: 1. Diehl et al. (2021) PLoS Pathogens 17:e1009969 2. Zhang et al. (2017) Sci Rep 7: 42188 3. Charnaud et al. (2017) PLoS One 12: e0181656 4. Külzer et al. (2012) Cell Micro 14: 1784-95 5. Külzer et al. (2010) Cell Micro 12: 1398-1420
