Projektübersicht
Dr. Stefan Rahlfs
Biochemie und Molekularbiologie
Justus-Liebig-Universität Gießen
Heinrich-Buff-Ring 26-32
35390 Gießen
Tel.: +49 (0)641-99 39117
E-Mail: stefan.rahlfs(at)ernaehrung.uni-giessen(dot)de

NAD(P)H-abhängige Stoffwechselwege als Ziele für neue Antiinfektiva
Projektbeschreibung
Das zelluläre Redoxgleichgewicht spielt für pathogene Mikroorganismen eine essentielle Rolle. Zentral daran beteiligt sind Enzyme des NAD(P)H-abhängigen Glutathion- und Thioredoxinsystems1,2, sowie die Glucose-6-Phosphatdehydrogenase (G6PD) und 6-Phosphogluconatdehydrogenase (6PGD), die maßgeblich zur Bereitstellung von NADPH und Ribose-5-Phosphat über den Pentosephosphatweg beitragen3. Die bifunktionale G6PD der Malariaparasiten Plasmodium falciparum und P. vivax (GluPho) unterscheidet sich funktionell und strukturell von den humanen Wirtsenzymen4 und ist essenziell für den Malariaparasiten5. In Kooperation mit dem Sanford-Burnham Institute/UCSD, La Jolla, konnten wir für die PfGluPho einen high-throughput kompatiblen Assay etablieren und ca. 400.000 Substanzen (u.a. NIH MLSMR Collection) screenen6. Anschließende Struktur-Wirkungs-Analysen und Lead-Optimierung resultierten in den im nanomolaren Bereich aktiven und hoch selektiven PfGluPho Inhibitor SBI-7507. Das Konzept konnte bereits auf Leishmania übertragen werden, die 3D-Kristallstruktur der Leishmania donovani G6PD und PGD gelöst werden und auf deren Basis ein erstes in silico Screening von Kleinmolekülen durchgeführt werden. Darüber hinaus wurde ein high-throughput kompatibler Assay für die rekombinanten G6PDs und 6PGDs aus Leishmania etabliert, um ein Inhibitor-Screening bei dem Novartis FAST lab in Cambridge (USA) durchzuführen.
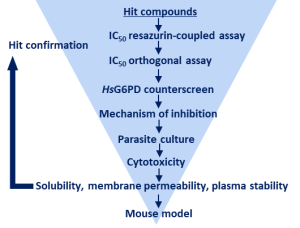
Workflow-Diagramm zur Inhibitor Identifizierung gegen G6PDs/PGDs
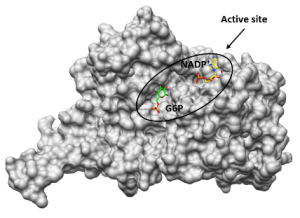
3D-Kristallstruktur LdG6PD ©Isabell Berneburg
Wissenschaftliches Ziel:
Ziel des Projektes ist die funktionelle und strukturelle Charakterisierung der Enzyme G6PD und 6PGD aus Leishmania und Plasmodium als Zielmoleküle für die Wirkstoffentwicklung, sowie die Identifizierung und Weiterentwicklung von Inhibitoren dieser Enzyme (in silico und HTS). Das Konzept soll auch auf weitere Erreger innerhalb des DRUID Konsortiums, wie z.B. Schistosoma, übertragen werden.
DRUID-Kooperationspartner:
B1 Diederich/Kolb; B4 Grevelding; D3 van Zandbergen; E1 Grevelding/Häberlein; E3 Rahlfs/Przyborski; E4 Spengler; E6 Schiffmann/Laux
Literatur B3: 1. Fritz-Wolf et al. (2011) Nature Comm. 2:383* 2. Koncarevic et al. (2009) PNAS 106: 13323-8* 3. Bozdech and Ginsburg (2005) Malaria J 3:23 4. Jortzik et al. (2011) Biochem J Energy 436:641-50* 5. Allen et al. (2015) FEBS J 282:3808-23*, 6. Preuss et al. (2012) J Med Chem 55:7262-72* 7. Berneburg et al. (2022) Antimicrob Agents Chemother (accepted)*
*eigene Publikationen
Proteinproduktion, HTS-Assays, Kristallisation, Interaktionsanalysen
Projektbeschreibung
Rahmen der Plattform [E3] sollen Methoden zur Proteinproduktion, zur Entwicklung von high-throughput Assays, zur Kristallisation und Strukturanalyse sowie zu Interaktionsanalysen für das DRUID-Konsortium bereitgestellt werden. Dabei kann eine Vielzahl an Optimierungsmöglichkeiten bei der Klonierung/Expression sowie bei der Reinigung nativer Proteine (Affinitäts-chromatographie, Gelfiltration, Ionenaustauscher) angeboten werden. Für erfolgreich rekombinant produzierte Proteine kann über die Plattform [E3] Unterstützung bei der Entwicklung von Assaysystemen bis hin zu high throughput-fähigen Formaten gegeben werden. Des Weiteren soll über das Projekt [E3] Zugang zu der Kristallisationsplattform am iFZ ermöglicht werden. Für Kristallisationscreens im nl-Massstab stehen ein Pipettier- sowie ein Kristallisationsroboter sowie Starterkits und diverse Additivscreens zur Optimierung zur Verfügung, z. Z. wird das System durch Finanzierung von DRUID erneuert. Frau Dr. Fritz-Wolf wird als Kristallografin und Ansprechpartnerin fungieren und kann die ersten Testungen der Kristalle vornehmen. Hierzu stehen die Röntgenquellen des Max-Planck-Instituts für Med. Forschung (Heidelberg) bzw. der AG Klebe/Heine [A5] (Marburg) zur Verfügung.
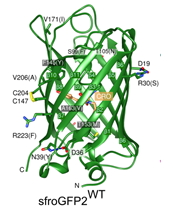
Struktur von sfroGFP (Heimsch et al. 2022)
DRUID-Kooperationspartner:
Im Bereich Protein-Expression: A1, A3, A6NWG, A7, B2, B3, B4, C3
Im Bereich Kristallisation: B2, B3, C3
Im Bereich Assay-Entwicklung: A7, B3
Literatur E3: 1. Heimsch et al. (2022) Antioxid & Redox Signal doi: 10.1089/ars.2021.0234 2. Harnischfeger et al. (2020) Electronic Journal of Biotechnology 3. Fritz-Wolf et al. (2011) Nat Commun 2:383 4. Koncarevic et al. (2009) Proc Natl Acad Sci USA 106:13323-8
